« Choisir les sondes pour tester SNP sur micro-array » : différence entre les versions
Aller à la navigation
Aller à la recherche
m (ajouté image de l'accès au SNP) |
mAucun résumé des modifications |
||
| Ligne 21 : | Ligne 21 : | ||
== Scénarios pédagogiques où il peut s'intégrer == | == Scénarios pédagogiques où il peut s'intégrer == | ||
* | |||
===== Références ===== | |||
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB | |||
Retour à [[Bioinformatique : opportunités pour l’enseignement]] | Retour à [[Bioinformatique : opportunités pour l’enseignement]] | ||
[[Catégorie: BioInfoScenarios]] | [[Catégorie: BioInfoScenarios]] | ||
Version du 1 octobre 2021 à 16:39
Savoir choisir les sondes pour déterminer chaque SNP étudié dans une puce à ADN (µ-array)
Procédure
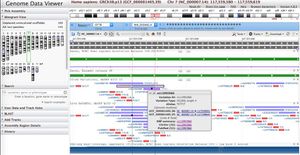
Ouvrir Genome data Viewer GDV ici
Prenez un exemple concret à partir d'un SNP par exemple dans le gène CFTR
Trouver le gène CFTR voir ici Séquence_du_gène_de_protéines_sur_chromosomes
si nécessaire zoomer à la séquence, choisir un snp par exemple rs1228922204 selon ce protocole SNP_fréquents_-_changements_d'une_seule_base
Noter la séquence de référence et celle du SNP (quelques bases de chaque coté pour un total de 25 environ)
-> comment établir des sondes pour le Micro-array
NB: cette procédure simplifiée pour les élèves n'est pas complète : il faudrait encore vérifier que ces séquences sont effectivement uniques.
Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe
Scénarios pédagogiques où il peut s'intégrer
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB