Preuve de l'évolution par la comparaison de protéines chez différentes espèces
Preuve de l'évolution par comparaison des séquences de protéine chez différentes espèces
Procédure
Trouver les séquences de la protéine CFTR chez plusieurs espèces dans la banque de données UniProtKB
UniProtKB est une banque de données qui contient toutes les séquences de protéine répertoriées à partir des données publiques (~230 millions de protéines).
N.B. : pour étudier l'évolution, on utilise souvent des séquences de protéine : elles sont plus pertinentes du point de vue évolutif. En effet elles déterminent en grande partie le phénotype, sur lequel la sélection s'effectue.
Cet exemple illustre la procédure avec la protéine CFTR (qui cause la mucoviscidose quand elle est défectueuse) mais d'autres protéines peuvent être employées (cf liste d'exemples)
- Aller sur UniProtKB dans Query taper gene:CFTR. Il faut chercher les protéines par leur nom du gène (1)
UniProt va chercher toutes les entrées dans lesquelles on trouve le terme CFTR solution (en date du 22.02.24): la liste est énorme.
- Sélectionner les entrées "reviewed" solution (Insuline) (en date du 23.09.22), solution CFTR(en date du 23.09.22)
(1) N.B. : Le nom de la protéine (cf liste d'exemples) diffère souvent du nom du gène. Il vaut mieux chercher une protéine par le nom de son gène, car les noms de gène sont 'standardisés' par des comités d'experts. UniProtKB est une banque de données sur les protéines : les informations sont focalisées sur les séquences de protéine mais la liste des noms de gènes est exhaustive. Un même gène peut parfois avoir plusieurs noms. Exemple: le gène CFTR a un synonyme : abcc7
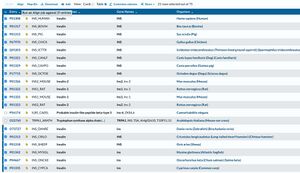
Sélectionner la même protéine chez plusieurs organismes
Sélectionner (cocher) dans la liste les protéines des espèces qui vous intéressent.
Un minimum de 5 séquences de protéine est nécessaire si l'on veut construire un alignement de séquences qui fait du sens. (cf liste d'exemples).
Il peut être judicieux d'imposer à chaque groupe quelques espèces et les laisser en choisir quelques autres à volonté, pour faciliter les comparaisons. P. ex. :
- humain,
- chimpanzé,
- souris,
- rat,
- bovin,
- cheval,
- Danio rero ( poisson),
- drosophile (pas disponible pour la protéine CFTR)
NB: Vérifier que le nom de gène affiché dans la liste est bien celui recherché et que les séquences de protéines sélectionnées sont de longueur similaire (parfois les chercheurs ont déterminé la séquence d'une partie de la protéine seulement - en fonction de leur étude). Ce n'est pas impossible, mais moins visuellement frappant et plus complexe à interpréter si on a une séquence bien plus courte ou plus longue.
Les séquences sélectionnées sont marquée d'un rectangle bleu en début de ligne et leur nombre apparaît dans le bandeau bleu en haut "21 rows selected out of 97" .
- Avant de passer à une autre page suivante cliquer 'Add' avec l’icône d'un panier, sinon la sélection est perdue.
N.B: si on avait sélectionné d'autres séquences auparavant : il faut cliquer 'Clear' dans ce bandeau pour éviter qu'elles restent sélectionnées.)
Construire un alignement
NB : Demander aux élèves d'effectuer une / des captures d'écran pour retrouver les noms d'espèces et pas seulement le nom d'entrée (INS_PANTR -> Pan troglodytes (Chimpanzee))
- Cliquer le bouton "Align" en-dessus du bandeau en haut de la liste. Une fenêtre affiches les séquences qui seront alignées au format FASTA.
- Cliquer le bouton en bas à droite "Align XX sequences". En principe 1 ne pas modifier les paramètres.
- Après un certain temps (plusieurs secondes, voire minutes) une fenêtre "Tool results" apparait affichant cet alignement et les alignements effectués auparavant dans la session.
- Cliquer "Completed" pour afficher l'alignement dans une nouvelle page. Exemple pour CFTR (valable 7 jours depuis le 23 09 22)

Les séquences alignées sont affichées sous forme de tableau une séquence (espèce) par ligne, 66 par ligne. (En mode Overview = par défaut)
Sur une ligne en-dessous, le signal de la protéine est indiqué en rouge.
NB: "-" (tiret) signifie que le programme d'alignement a introduit un espace (insertion/délétion ou "gap") pour pouvoir aligner des séquences de différentes longueurs.
1 On peut toutefois modifier la première ligne pour chaque espèce afin de mettre le nom d'espèce en premier plutôt que le code : par exemple
>sp|P67970|INS_CHICK OS=Gallus gallus OX=9031 GN=INS PE=1 SV=1
>sp|P67970|Gallus gallus OX=9031 GN=INS PE=1 SV=1
Sinon Pour retrouver le nom de l'espèce, cliquer sur le numéro d'accession en bleu -> une nouvelle fenêtre s'ouvre avec l'entrée UniProtKB correspondant à cette protéine et le nom complet de l'espèce.
Pour aller plus loin
Le highlight "Similarity" en dessus du tableau à gauche est activé par défaut : les régions similaires sont mises en évidence (violet) - permettant de discuter celles qui sont évolutivement plus conservées.
- Highlighter "Physical properties" puis "Hydrophobicity" : met en évidence des régions susceptibles d'être transmembranaires
Une matrice des degrés de similitude est accessible en choisissant
Compléments
Les propriétés physico-chimiques des différents acides aminés sont décrites ici Source : M.-C. Blatter.
Un tableau de correspondance des codes à 3 lettres et à1 lettre des acides aminés et des codons est accessible ici Source : Leder et al. Introduction to molecular medicine Ed Sientific American (94)
Les taux de similitude en %
Une matrice des degrés de similitude est accessible en choisissant percent identity matrix exemple .
On peut imaginer des activités avec les données numériques - par exemple en collaboration avec le cours de mathUn arbre … phylogénétique ?
En haut de la page , au lieu de la visualisation "overview" on peut cliquer "Trees" pour afficher des arbres avec différentes visualisations... MAIS l'arbre n'est pas phylogénétique : c'est un guided tree, utilisé par le programme pour construire l'alignement et est basé uniquement sur les différences observées entre les séquences. Il ne s'agit pas d'un arbre phylogénétique: un arbre phylogénétique est beaucoup plus complexe à établir ! Voir par exemple le scénario SIB-3. Phylogénie, biodiversité et pizza ...
Voir aussi Trouver la date de divergence évolutive de deux espèces
Des questions possibles :
- Trouver les organismes les plus proches de (humain, souris, vache, ...) pour chacune des protéines étudiées par la classe. Essayer de trouver une autre explication que l’origine commune et l'évolution indépendante depuis la séparation.
- Pourquoi utiliser les séquences ''protéiques'' plutôt que nucléotides dans ce contexte évolutif ?
- Y a-t-il des séquences qui sont très similaires chez un grand nombre d'organismes ?
- Constater que les séquences de l'insuline, de l'EPO, d'une Histone, de CFTR, etc. sont très similaires chez de nombreuses espèces : on peut aider les élèves à y voir l'expression de cette unicité fondamentale?
- L'insuline varie dans bien des régions, alors que l'Histone (H4) est presque identique entre de très nombreux organismes.On peut discuter la pression de sélection qui a éliminé tous les organismes dont l'histone n'était pas à même de remplir ses fonctions dans l'enroulement et la régulation de l'ADN.
- Est-ce que des séquences similaires entre un grand nombre d'organismes indiquent qu'il n'y a pas de mutations à ces endroits-là ?
- On peut aussi noter que la discussion sur les alignements permet de mieux mettre en évidence les concepts fondamentaux d'origine commune et de degré de similarité coordonnés entre diverses protéines
- Demander aux élèves de rechercher l'époque où se sont séparés les espèces étudiées et comparer avec l'arbre obtenu. Cf. Trouver la date de divergence évolutive de deux espèces
Reférences
Scénario établi sur la base des indications scientifiques de M.-C. Blatter du SIB