Gènes constitués d'Introns et d'Exons
Aller à la navigation
Aller à la recherche
Éprouver que certains gènes sont constitués d'Introns et d'Exons
Procédure
- Ouvrir Genome data Viewer GDV ici
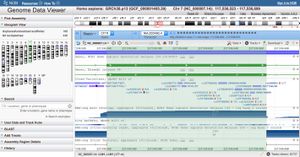
- Choisir l'espèce humaine
- Sélectionner un gène (p. ex CFTR) selon scénario trouver séquence des gènes d'une protéine (solution)
- Cliquer les petits ronds dans la bande bleue pour mettre en évidence un exon à la fois .
- Observer la séquence d'ADN correspondante et le sens de lecture (flèches dans la bande verte)
- Observer les bandes bleues plus bas sous RNAseq exon coverage et RNAseq intron spanning read
- NB. Si on a dézoomé trop, un message avertit qu'il a trop d'exons à afficher " Exon navigator; there are too many exons (number) in the region: please narrow the region to enable exon navigation"
- Recommencer avec 3-4 exons pour observer leur position, leur ampleur sur le gène
Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe
Synthèse d'un élève : En zoomant sur le gène en question ( cela fonctionne pour tous les gènes étudiés dans la classe, on remarque la présence d’introns et d’exons à l’aide des blocs et lignes colorées en verts ( ou différentes couleurs ). Les exons, plus petits que les introns sont aussi moins exprimés que ces introns. La présence d’introns prouve l’utilisation de l’épissage alternatif de la part du gène.
Scénarios pédagogiques où il peut s'intégrer
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB