« Preuve de l'évolution par la comparaison de protéines chez différentes espèces » : différence entre les versions
Aucun résumé des modifications |
m (contiunué à rédiger) |
||
| Ligne 20 : | Ligne 20 : | ||
=== 2° Afficher les données pertinentes === | === 2° Afficher les données pertinentes === | ||
Cliquer "Customize display" et sélectionner les données dont on souhaite l'affichage : p. ex. Organism, Protein Names, Gene Names, Sequence. | Cliquer "Customize display" et sélectionner les données dont on souhaite l'affichage : p. ex. Organism, Protein Names, Gene Names, Sequence. | ||
[[Fichier:Uniprot-selected-ins-div-sp.jpg|alt=sélection de protéines dans unprot KB|vignette]] | |||
=== 3° Sélectionner la même protéine chez plusieurs organismes === | |||
=== 3° Sélectionner la même protéine chez plusieurs organismes | |||
Sélectionner (cocher) dans cette liste les insulines des espèces qui vous intéressent. Un minimum de 5 est nécessaire si l'on veut ensuite former un alignement de séquences raisonnable. | Sélectionner (cocher) dans cette liste les insulines des espèces qui vous intéressent. Un minimum de 5 est nécessaire si l'on veut ensuite former un alignement de séquences raisonnable. | ||
Les séquences | Les séquences son surlignées de jaune et leur nombre dans le bandeau jaune en haut. | ||
(N.B: si on avait sélectionné d'autres séquences | (N.B: si on avait sélectionné d'autres séquences auparavant : il faut cliquer Clear dans ce bandeau pour éviter qu'elles restent sélectionnées.) | ||
=== 4° Produire un alignement === | === 4° Produire un alignement === | ||
Cliquer le bouton Align | Cliquer le bouton Align en-dessus du bandeau en haut de la liste. Après un temps de plusieurs secondes en général l'alignement est affiché | ||
[[Fichier:Alignement-ins-similarity.jpg|alt=Alignement de quelques insulines de diverses espèces - similarité activée |vignette|Alignement de quelques insulines de diverses espèces - similarité activée ]] | |||
Cocher la case "Similarity" dans la colonne de gauche | |||
les séquences alignées sont affichées et on peut observer que certaines zones sont plus conservées. | |||
Une étoile "*" signifie identité ( dans la colonne) pour toutes les séquences alignées. ":" signifie acides aminées qui ont des propriétés physico-chimiques '''très''' similaires, "." signifie acides aminées qui ont des propriétés physico-chimiques similaires, un "-" signifie que le le programme d'alignement a introduit un espace ("gap") pour aligner avec des séquences plus longues | |||
Les propriétés chimiques des différents a.a. sont décrites ici | |||
Un tableau des codes à 3 lettres et à1 lettre et codons. | |||
Pour pouvoir retrouver les noms d'espèce on peut imprimer ou copier-coller le tableau - plus haut dans la même page - donnant les noms des espèces en rapport avec le numéro d'accession. | |||
== ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | == ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | ||
Version du 26 mars 2020 à 19:02
Preuve de l'évolution par la comparaison de séquences de protéines chez différentes espèces
Procédure
1Trouver les entrées pour insuline de plusieurs espèces dans la banque de données UniProtKB
N. B. : En principe on utilise les séquences protéiques : elles sont plus pertinentes du point de vue évolutif (phénotype). Ici nous travaillons avec l'insuline, mais d'autres protéines peuvent être employées (cf liste d'exemples)
Aller sur UniProtKB dans Query taper insulin. Si on écrit en français il propose "Did you mean: insulin ": cliquer le terme en anglais.
UniProtKB contient toutes les protéines répertoriées à partir des données publiques (~180 millions début 2020).
va chercher toutes les entrées dans lesquelles on trouve le terme insulin solution : la liste est énorme.
Cliquer Restrict term "insulin" to ....Protein name et cliquer Filter by puis "reviewed" solution,
N.B. Le nom de la protéine (cf liste d'exemples) diffère souvent du nom du gène. Il faut parfois chercher par le nom du gène pour retrouver le nom de la protéine, ou l'inverse. M . UniProtKB est une banque de données sur les protéines : les informations sont focalisées sur les séquences protéiques mais la liste des noms de gènes est toutefois exhaustive.
En haut de la liste : l'entrée pour l'insuline humaine INS_HUMAN : P01308 Solution.
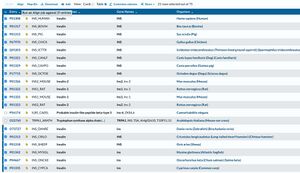
2° Afficher les données pertinentes
Cliquer "Customize display" et sélectionner les données dont on souhaite l'affichage : p. ex. Organism, Protein Names, Gene Names, Sequence.
3° Sélectionner la même protéine chez plusieurs organismes
Sélectionner (cocher) dans cette liste les insulines des espèces qui vous intéressent. Un minimum de 5 est nécessaire si l'on veut ensuite former un alignement de séquences raisonnable.
Les séquences son surlignées de jaune et leur nombre dans le bandeau jaune en haut.
(N.B: si on avait sélectionné d'autres séquences auparavant : il faut cliquer Clear dans ce bandeau pour éviter qu'elles restent sélectionnées.)
4° Produire un alignement
Cliquer le bouton Align en-dessus du bandeau en haut de la liste. Après un temps de plusieurs secondes en général l'alignement est affiché
Cocher la case "Similarity" dans la colonne de gauche
les séquences alignées sont affichées et on peut observer que certaines zones sont plus conservées.
Une étoile "*" signifie identité ( dans la colonne) pour toutes les séquences alignées. ":" signifie acides aminées qui ont des propriétés physico-chimiques très similaires, "." signifie acides aminées qui ont des propriétés physico-chimiques similaires, un "-" signifie que le le programme d'alignement a introduit un espace ("gap") pour aligner avec des séquences plus longues
Les propriétés chimiques des différents a.a. sont décrites ici
Un tableau des codes à 3 lettres et à1 lettre et codons.
Pour pouvoir retrouver les noms d'espèce on peut imprimer ou copier-coller le tableau - plus haut dans la même page - donnant les noms des espèces en rapport avec le numéro d'accession.