« Éprouver le lien entre structure, séquence et fonction d'une protéine » : différence entre les versions
mAucun résumé des modifications |
Aucun résumé des modifications |
||
| Ligne 4 : | Ligne 4 : | ||
* Choisir une protéine et aligner les séquences pour plusieurs espèces et aligner les protéines selon ce scénario [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]] | * Choisir une protéine et aligner les séquences pour plusieurs espèces et aligner les protéines selon ce scénario [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]] | ||
* Chercher la protéine p. ex. [https://www.uniprot.org/uniprot/?query=gene%3Acftr&sort=score CFTR] sur le site UniProt?, on peut aligner la séquence d'acide aminées et la comparer entre différentes espèces animales (ici l'humaine, la souris, le rat, le chien, un cochon d'Asie et le lapin). | * Chercher la protéine p. ex. [https://www.uniprot.org/uniprot/?query=gene%3Acftr&sort=score CFTR] sur le site UniProt?, on peut aligner la séquence d'acide aminées et la comparer entre différentes espèces animales (ici l'humaine, la souris, le rat, le chien, un cochon d'Asie et le lapin). | ||
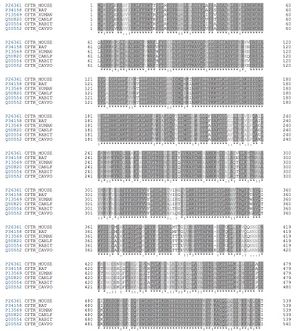
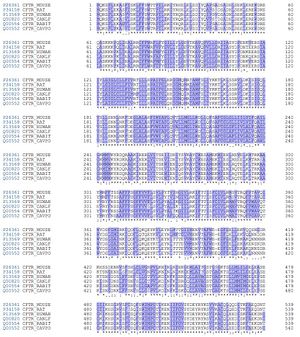
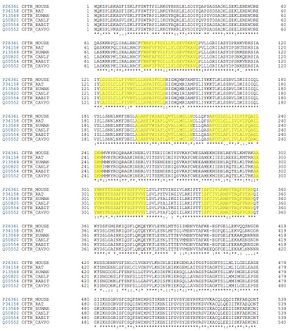
* Cocher "Similarity" puis "Hydrophobic" puis transmembrane[[Fichier:CFTR-alignement-similarity-narrow.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec similarity activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec similarity activé]][[Fichier:CFTR-alignement-hydrophobic.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec Hydrophobic activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec Hydrophobic activé]][[Fichier:CFTR-alignement-transmembrane-narrow.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec transmembrane activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec transmembrane activé]] | * Cocher "Similarity" puis "Hydrophobic" puis transmembrane | ||
<gallery widths=50px heights=50px> | |||
Image:Greenway_model.gif|Greenway model | |||
Image:Greenway_model.gif|Greenway model | |||
</gallery> | |||
[[Fichier:CFTR-alignement-similarity-narrow.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec similarity activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec similarity activé]][[Fichier:CFTR-alignement-hydrophobic.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec Hydrophobic activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec Hydrophobic activé]][[Fichier:CFTR-alignement-transmembrane-narrow.jpg|alt=Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec transmembrane activé|vignette|Résultats d'un alignement de la séquence pour plusieurs espèces CFTR dans UniProt avec transmembrane activé]] | |||
* Certaines portions de la séquence sont identiques (en gris foncé) pour toutes les espèces, ce qui pourrait vouloir dire dire qu'une mutation sur ces endroits de la séquence de la protéine ne permettrait pas la formation d'une protéine fonctionnelle et on ne pourrait donc pas voir de protéines comportant une mutations à ces endroits. Il est aussi fort probable que ces portions identiques de la séquence soient le site actif de la protéine, et qu'une mutation rendrait donc la protéine inactive. | * Certaines portions de la séquence sont identiques (en gris foncé) pour toutes les espèces, ce qui pourrait vouloir dire dire qu'une mutation sur ces endroits de la séquence de la protéine ne permettrait pas la formation d'une protéine fonctionnelle et on ne pourrait donc pas voir de protéines comportant une mutations à ces endroits. Il est aussi fort probable que ces portions identiques de la séquence soient le site actif de la protéine, et qu'une mutation rendrait donc la protéine inactive. | ||
De plus, on observe également que les portions de la séquence d'acide aminées qui sont hydrophobes sont presque toutes identiques pour tous les espèces. Cela s'explique par le fait que la protéine est une protéine de transport trans-membranaire du chlore. S'il y a des mutations sur les portions de la séquence qui sont hydrophobes, et donc que la structure de ces portions ne traverse plus la membrane, il est probable que la protéine ne soit plus efficace du fait que sa fonction de transport trans-membranaire ne puisse plus être remplie. | De plus, on observe également que les portions de la séquence d'acide aminées qui sont hydrophobes sont presque toutes identiques pour tous les espèces. Cela s'explique par le fait que la protéine est une protéine de transport trans-membranaire du chlore. S'il y a des mutations sur les portions de la séquence qui sont hydrophobes, et donc que la structure de ces portions ne traverse plus la membrane, il est probable que la protéine ne soit plus efficace du fait que sa fonction de transport trans-membranaire ne puisse plus être remplie. | ||
Version du 28 septembre 2020 à 11:06
Éprouver le lien entre structure, séquence et fonction d'une protéine
Procédure
- Choisir une protéine et aligner les séquences pour plusieurs espèces et aligner les protéines selon ce scénario Preuve de l'évolution par la comparaison de protéines chez différentes espèces
- Chercher la protéine p. ex. CFTR sur le site UniProt?, on peut aligner la séquence d'acide aminées et la comparer entre différentes espèces animales (ici l'humaine, la souris, le rat, le chien, un cochon d'Asie et le lapin).
- Cocher "Similarity" puis "Hydrophobic" puis transmembrane
- Certaines portions de la séquence sont identiques (en gris foncé) pour toutes les espèces, ce qui pourrait vouloir dire dire qu'une mutation sur ces endroits de la séquence de la protéine ne permettrait pas la formation d'une protéine fonctionnelle et on ne pourrait donc pas voir de protéines comportant une mutations à ces endroits. Il est aussi fort probable que ces portions identiques de la séquence soient le site actif de la protéine, et qu'une mutation rendrait donc la protéine inactive.
De plus, on observe également que les portions de la séquence d'acide aminées qui sont hydrophobes sont presque toutes identiques pour tous les espèces. Cela s'explique par le fait que la protéine est une protéine de transport trans-membranaire du chlore. S'il y a des mutations sur les portions de la séquence qui sont hydrophobes, et donc que la structure de ces portions ne traverse plus la membrane, il est probable que la protéine ne soit plus efficace du fait que sa fonction de transport trans-membranaire ne puisse plus être remplie.
| |
Figure 4 : Alignement des séquences d'acides aminées synthétisées par le gène CFTR avec en gris les zones de similarité entre les différents organismes et en violet les zones hydrophobes de la protéine associée