Trouver des séquences virales dans le génome humain
Trouver des séquences virales dans le génome humain
Concept pédagogique
Notre ADN est truffé de séquences d'autres organismes (virales notamment mais aussi bactérienne, etc.) :
a) Certains gènes humains (nécessaires ) sont d'origine virale (les HERV dont ERVW1)
b) ce gène se retrouve chez plusieurs autres espèces mais d) seulement proches phylogénétiquement
Les faire trouver par les élèves peut les aider à éprouver l'ampleur des transferts horizontaux, la grande perméabilité de notre ADN à travers le temps, sur les mécanismes qui maintiennent l'unicité des espèces malgré les forces divergentes.
Cela pourrait aussi fonder la discussion sur les effets possibles d’absorber des gènes étrangers dans le débat sur les OGM
d) L'analyse du génome humain a révélé, entre autres, que 8 à 10 % de la séquence du génome est d'origine virale ! ...alors que seulement ~1.5 % de la séquence du génome 'code' pour des protéines !
Les séquences d'ADN virales sont des vestiges de l'infection, datant de plusieurs millions d'années, des cellules germinales de nos ancêtres primates par des virus appelés 'rétrovirus'. La plupart de ces séquences sont inactives: elles ont subi des modifications les rendant incapables de coder pour la moindre protéine.
Procédure
Rétrovirus endogènes produisant des protéines
Complément d'info : Certaines séquences de rétrovirus endogènes sont 'actives' et produisent des protéines 'fonctionnelles' (Exemple les rétrovirus HERV -> gène ERVW1)
Liste des protéines humaines connues produites à partir des séquences des rétrovirus endogènes 'HERV' dans UniProtKB.
Complément d'info : Parmi les protéines humaines produites par des séquences virales intégrées dans le génome humain, on trouve les syncytines (syncytin).
Ces protéines ont des propriétés fusiogènes (vestiges des gènes 'env') et joueraient un rôle dans la formation du placenta. Elles auraient également des propriétés immunosuppressives, essentielles pour une interface mère-enfant (Dossier La Recherche XI 010).
a) Localiser le gène de la syncytine dans le génome humain
Localiser le gène de la syncytine (appelé ERVWE1) sur le génome humain à l'aide de GDV (selon scénario ici)
- Résultat: Le gène codant pour la syncytine (ERVWE1) se trouve sur le chromosome 7 solution ici
b) Trouver les protéines similaires à la syncytine humaine chez d'autres espèces
Complément d'info : BLAST permet de comparer la séquence d'une protéine avec des millions d'autres séquences contenues dans une banque de données et de retrouver celles qui lui ressemblent le plus.
A partir de l'entrée UniProtKB correspondant à la syncytine humaine Q9UQF0 Syncytin humaine
- Cliquer sur l'onglet BLAST
- Cliquer sur 'Default'
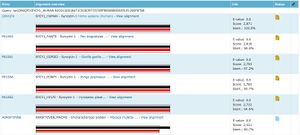
- Cliquer sur le bouton 'Blast' BLAST trouve plusieurs gènes chez d'autres espèces avec une E-value très basses (cf donc des similitudes très significatives : cf figure ci-contre
Complément d'info : Le rétrovirus à l'origine des syncytines aurait été 'intégré' par un ancêtre primate, il y a 45 à 70 millions d'années. La syncytine n'est retrouvée que chez les descendants de ces primates (les simiens).
- Trouver les protéines virales les plus similaires à la syncytine humaine et notez le nom de l'espèce concernée.
Complément d'info : L'ancêtre du gène de la syncytine est le gène viral 'env'. Les syncytines ont 'récupéré' les propriétés fusiogènes des gènes viraux 'env'.
- Voir sur UniProt liste des protéines humaines codées par des séquences virales ici
c) Comparer les séquences des protéines similaires à la syncytine humaine
- A partir des résultats du Blast sur UniProt
- Sélectionner des protéines similaires de différentes espèces (mammifères et virus)
- Dans la barre verte qui apparaît en bas de l'écran, cliquez sur 'Clear, puis sur 'Align'
- Visualiser ainsi les différences et les similitudes entre les différentes séquences en acides aminés.
- Pour mieux voir les similarités: cliquer sur 'Similarity'.
Exemples:
- Comparer (aligner) les séquence de l'homme et du chimpanzé
- Comparer (aligner) les séquence de l'homme et du Baboon endogenous virus
d) Localiser des séquences d'origine virale dans le génome humain Ouvrir BLAT (un autre genome Viewer) ici iOn peut visualiser les zones répétées dans un chromosome - ici une partie du 8 considérer les LTR seulement .
- Griffiths, D. J. (2001). Endogenous retroviruses in the human genome sequence. Genome Biology, 2(6),https://dx.doi.org/10.1186%2Fgb-2001-2-6-reviews1017
Voir aussi
- Virusite : recense et permet d'accéder aux génomes de virus
- p. ex. pour SARS-CoV-2 :
- on accède à la Séquence format FASTA et sa visualisation graphique dans GDV nsp est non-structural protein
- L'ensemble des séquences actuellement répertoriées : cliquer FASTA ou GRAPHICS pour visualiser dans GDV
- L'ensemble des protéines exprimées du SARS-CoV-2 sur UniProt
- p. ex. pour SARS-CoV-2 :
- Activité Coronavirus et protéine @SIB
Scénarios pédagogiques où il peut s'intégrer
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB (voir http://education.expasy.org/bioinformatique/Human_virus_bacteria.html)