« Etablir l'alignement et une phylogénie » : différence entre les versions
Aucun résumé des modifications |
Aucun résumé des modifications |
||
| Ligne 3 : | Ligne 3 : | ||
== Procédure == | == Procédure == | ||
Suite à une publication récente (Lemopoulos & Montoya‐Burgos , 2021) | Suite à une publication récente (Lemopoulos & Montoya‐Burgos , 2021) sur l'évolution des écailles, des plaques osseuses ou une peau nue, des écailles, sur la base d'une phylogénie établie par la comparaison bioinformatique des séquences. Cf [https://jump-to-science.unige.ch Jump-To-Science] ''lien sur la publication a ajouter ici'' . Juan Montoya‐Burgos a sélectionné pour le projet [https://jump-to-science.unige.ch/ Jump-To-Science] quelques séquences parmi les milliers utilisées dans la publication, pour un gène qui ne possède qu'une copie par espèce (single copy gene), qui montre suffisamment de variation et qui n'est pas trop difficile à aligner. | ||
Il vous a donc sélectionné chez plusieurs espèces de poissons la séquences du gène qui code pour: ''Cilia and flagella associated protein 5''8. On trouve cette protéine CFA58 pour de nombreuses espèces sur [https://www.uniprot.org/uniprot/?query=cilia+name%3Aflagella+associated+58&sort=score Uniprot : sélection ici] , même chez l'humain [https://www.uniprot.org/uniprot/Q5T655 Q5T655] | |||
===== Obtenir les séquences ===== | |||
* | * Downloader le [https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Cilia_associated_prot58_poissons.txt fichier texte ici] qui content les séquences non-alignées pour quelques 21 poissons et le requin (groupe externe), en format FASTA (Le code après le nom des espèces correspond à l'ID du gène dans la base de donnée [https://www.ncbi.nlm.nih.gov/genbank/ GenBank] | ||
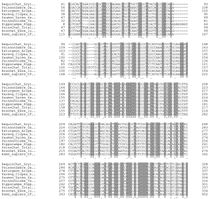
[[Fichier:Alignment-22-especes-poisson-Uniprot-similarity.jpg|alt=Alignment de 22-espèces de poissons sur Uniprot similarity on|vignette|141x141px|Alignment de 22-espèces de poissons sur Uniprot similarity on]] | [[Fichier:Alignment-22-especes-poisson-Uniprot-similarity.jpg|alt=Alignment de 22-espèces de poissons sur Uniprot similarity on|vignette|141x141px|Alignment de 22-espèces de poissons sur Uniprot similarity on]] | ||
===== Aligner | ===== Aligner ces séquences ===== | ||
* Ouvrir | * Ouvrir [https://uniprot.org UniProt] | ||
* Choisir [https://www.uniprot.org/align/ Align] | * Choisir [https://www.uniprot.org/align/ Align] | ||
* Coller toutes le [https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Cilia_associated_prot58_poissons.txt texte avec les séquences] dans le champ indiqué Protéin sequences (FASTA) | * Coller toutes le [https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Cilia_associated_prot58_poissons.txt texte avec les séquences] dans le champ indiqué Protéin sequences (FASTA) | ||
* Cliquer "Run align" | * Cliquer "Run align" | ||
* Après une attente variable de l'ordre de | * Après une attente variable de l'ordre de 1-3 minutes, on obtient un alignement (Exemple de résultat [https://www.uniprot.org/align/A20210505216DA2B77BFBD2E6699CA9B6D1C41EB200F9FCY ici] (actif jusqu'à mi juin 21) | ||
** Cliquer la case "Similarity" | |||
** Observer le grand degré de similarité - visible dans l'image ci-contre et [https://edutechwiki.unige.ch/fr/Fichier:Alignment-22-especes-poisson-Uniprot-similarity.jpg ici] | ** Observer le grand degré de similarité - visible dans l'image ci-contre et [https://edutechwiki.unige.ch/fr/Fichier:Alignment-22-especes-poisson-Uniprot-similarity.jpg ici] | ||
Noter l'arbre en dessous de l'alignement : "Tree" cf. pour la validité de cet arbre [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]]) | Noter l'arbre en dessous de l'alignement : "Tree" cf. pour une discussion de la validité de cet arbre cf ce scénario: [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]]) | ||
Un fichier PDF établi par Montoya‐Burgos montrant l'arbre que l'on obtient avec ce gène disponible [https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Arbre_ML_Cilia_associated_prote58_poissons_align-PhyML_tree.pdf ici] | Un fichier PDF établi par Montoya‐Burgos montrant l'arbre que l'on obtient avec ce gène et disponible [https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Arbre_ML_Cilia_associated_prote58_poissons_align-PhyML_tree.pdf ici] | ||
Il est intéressant de voir que les espèces Hippocampe et PoissonGlobe évoluent plus vite pour ce gène que les autres espèces).[https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Arbre_ML_Cilia_associated_prote58_poissons_align-PhyML_tree.jpg ici] | Il est intéressant de voir que les espèces Hippocampe et PoissonGlobe évoluent plus vite pour ce gène que les autres espèces).[https://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Arbre_ML_Cilia_associated_prote58_poissons_align-PhyML_tree.jpg ici] | ||
===== Pour aller plus loin ===== | ===== Pour aller plus loin ===== | ||
Pour éprouver le degré de similitude de ces espèces et discuter de l'origine commune de ces espèces (cf [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]]) | Pour éprouver le degré de similitude de ces espèces et discuter de l'origine commune de ces espèces (cf. [[Preuve de l'évolution par la comparaison de protéines chez différentes espèces]]) | ||
* | * Ajouter les séquences d'autres espèces dans la case "You may add additional sequences to this alignment (FASTA format) | ||
* Cliquer "add sequence and align" pour refaire l’alignement : exemple avec l'humain [http://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/ | **[[Fichier:Alignement-Cilia associated prot58-22poissons+homo-similarity-on.jpg|alt=Alignement dans Uniprot des séquences pour Cilia_associated_prot58 pour 22 poissons+homo avec Similarity on|vignette|210x210px|Alignement dans Uniprot des séquences pour Cilia_associated_prot58 pour 22 poissons+homo avec Similarity on]]On les obtient par exemple depuis le nom de la protéine sur genbank : pour [https://www.ncbi.nlm.nih.gov/gene?Db=gene&Cmd=DetailsSearch&Term=159686 Homo sapiens] on trouve une visualisation du gène similaire à Gene Data Viewer sous "Genomic regions, transcripts, and products" | ||
* Résultat : un extrait avec la similarité [http://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/alignement-simimilarity-poissons+homo.jpg ici | **Si on survole le nom du gène un menu offre l'accès - notamment [https://www.ncbi.nlm.nih.gov/protein/NP_001008723.1?report=fasta à la séquence en FASTA] ''ICI vérifier , ça n'a pas l'air facile'' | ||
* Rvenir dans Uniprot sur l'alignement et ajouter cette séquence en plus | |||
* Cliquer "add sequence and align" pour refaire l’alignement : exemple avec l'humain [http://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/Cilia_associated_prot58-homo-sap.txt ici''à corriger c'est cfp 36 par erreur''] | |||
* Résultat : image d'un extrait avec la similarité activée [http://tecfa.unige.ch/perso/lombardf/projets/experimental/sequences-poissons-V21/alignement-simimilarity-poissons+homo.jpg ici] | |||
[[Catégorie: BioInfoScenarios]] | [[Catégorie: BioInfoScenarios]] | ||
| Ligne 43 : | Ligne 41 : | ||
== Références == | == Références == | ||
Lemopoulos, A., & Montoya‐Burgos, J. I. (2021). From scales to armor : Scale losses and trunk bony plate gains in ray‐finned fishes. ''Evolution Letters'', evl3.219. https://doi.org/10.1002/evl3.219 | Lemopoulos, A., & Montoya‐Burgos, J. I. (2021). From scales to armor : Scale losses and trunk bony plate gains in ray‐finned fishes. ''Evolution Letters'', evl3.219. https://doi.org/10.1002/evl3.219 | ||
<span class="Z3988" title="url_ver=Z39.88-2004&ctx_ver=Z39.88-2004&rfr_id=info%3Asid%2Fzotero.org%3A2&rft_id=info%3Adoi%2F10.1002%2Fevl3.219&rft_val_fmt=info%3Aofi%2Ffmt%3Akev%3Amtx%3Ajournal&rft.genre=article&rft.atitle=From%20scales%20to%20armor%3A%20Scale%20losses%20and%20trunk%20bony%20plate%20gains%20in%20ray%E2%80%90finned%20fishes&rft.jtitle=Evolution%20Letters&rft.stitle=Evolution%20Letters&rft.aufirst=Alexandre&rft.aulast=Lemopoulos&rft.au=Alexandre%20Lemopoulos&rft.au=Juan%20I.%20Montoya%E2%80%90Burgos&rft.date=2021-03-23&rft.pages=evl3.219&rft.issn=2056-3744%2C%202056-3744&rft.language=en"></span> | |||
Retour à [[Bioinformatique : opportunités pour l’enseignement]]<span class="Z3988" title="url_ver=Z39.88-2004&ctx_ver=Z39.88-2004&rfr_id=info%3Asid%2Fzotero.org%3A2&rft_id=info%3Adoi%2F10.1002%2Fevl3.219&rft_val_fmt=info%3Aofi%2Ffmt%3Akev%3Amtx%3Ajournal&rft.genre=article&rft.atitle=From%20scales%20to%20armor%3A%20Scale%20losses%20and%20trunk%20bony%20plate%20gains%20in%20ray%E2%80%90finned%20fishes&rft.jtitle=Evolution%20Letters&rft.stitle=Evolution%20Letters&rft.aufirst=Alexandre&rft.aulast=Lemopoulos&rft.au=Alexandre%20Lemopoulos&rft.au=Juan%20I.%20Montoya%E2%80%90Burgos&rft.date=2021-03-23&rft.pages=evl3.219&rft.issn=2056-3744%2C%202056-3744&rft.language=en"></span> | |||
[[Catégorie: BioInfoScenarios]] | [[Catégorie: BioInfoScenarios]] | ||
Version du 6 mai 2021 à 16:34
Etablir l'alignement et une phylogénie à partir de données authentiques d'une publication récente
Procédure
Suite à une publication récente (Lemopoulos & Montoya‐Burgos , 2021) sur l'évolution des écailles, des plaques osseuses ou une peau nue, des écailles, sur la base d'une phylogénie établie par la comparaison bioinformatique des séquences. Cf Jump-To-Science lien sur la publication a ajouter ici . Juan Montoya‐Burgos a sélectionné pour le projet Jump-To-Science quelques séquences parmi les milliers utilisées dans la publication, pour un gène qui ne possède qu'une copie par espèce (single copy gene), qui montre suffisamment de variation et qui n'est pas trop difficile à aligner.
Il vous a donc sélectionné chez plusieurs espèces de poissons la séquences du gène qui code pour: Cilia and flagella associated protein 58. On trouve cette protéine CFA58 pour de nombreuses espèces sur Uniprot : sélection ici , même chez l'humain Q5T655
Obtenir les séquences
- Downloader le fichier texte ici qui content les séquences non-alignées pour quelques 21 poissons et le requin (groupe externe), en format FASTA (Le code après le nom des espèces correspond à l'ID du gène dans la base de donnée GenBank
Aligner ces séquences
- Ouvrir UniProt
- Choisir Align
- Coller toutes le texte avec les séquences dans le champ indiqué Protéin sequences (FASTA)
- Cliquer "Run align"
- Après une attente variable de l'ordre de 1-3 minutes, on obtient un alignement (Exemple de résultat ici (actif jusqu'à mi juin 21)
- Cliquer la case "Similarity"
- Observer le grand degré de similarité - visible dans l'image ci-contre et ici
Noter l'arbre en dessous de l'alignement : "Tree" cf. pour une discussion de la validité de cet arbre cf ce scénario: Preuve de l'évolution par la comparaison de protéines chez différentes espèces)
Un fichier PDF établi par Montoya‐Burgos montrant l'arbre que l'on obtient avec ce gène et disponible ici
Il est intéressant de voir que les espèces Hippocampe et PoissonGlobe évoluent plus vite pour ce gène que les autres espèces).ici
Pour aller plus loin
Pour éprouver le degré de similitude de ces espèces et discuter de l'origine commune de ces espèces (cf. Preuve de l'évolution par la comparaison de protéines chez différentes espèces)
- Ajouter les séquences d'autres espèces dans la case "You may add additional sequences to this alignment (FASTA format)
- On les obtient par exemple depuis le nom de la protéine sur genbank : pour Homo sapiens on trouve une visualisation du gène similaire à Gene Data Viewer sous "Genomic regions, transcripts, and products"
- Si on survole le nom du gène un menu offre l'accès - notamment à la séquence en FASTA ICI vérifier , ça n'a pas l'air facile
- Rvenir dans Uniprot sur l'alignement et ajouter cette séquence en plus
- Cliquer "add sequence and align" pour refaire l’alignement : exemple avec l'humain icià corriger c'est cfp 36 par erreur
- Résultat : image d'un extrait avec la similarité activée ici
Insertions possibles des activités de biologie numérique
Concepts et Scénarios pédagogiques où il peut s'intégrer
Références
Lemopoulos, A., & Montoya‐Burgos, J. I. (2021). From scales to armor : Scale losses and trunk bony plate gains in ray‐finned fishes. Evolution Letters, evl3.219. https://doi.org/10.1002/evl3.219