Tutoriel tm text mining package
| Tutoriels R | |
|---|---|
| ▲ | |
| ◀▬▬▶ | |
| ⚐ brouillon | ☸ intermédiaire |
| ⚒ 2014/12/03 | ⚒⚒ 2014/11/18 |
| Prérequis | |
| Voir aussi | |
| Sous-pages et productions: | |
Introduction
tm est un paquet (extension) R. C'est un "framework" pour l'analyse statistique de textes. Le logiciel aide pour tout ce qui est préparation, constitution de corpus, analyses simples (fréquences, etc.). On peut construire des tableaux de proximité que l'on peut ensuite analyser avec des outils statistiques.
Ce tutoriel montrera comment profiter de la plupart des fonctionalités du paquet tm. On s'intéressera aussi à l'interopérabilité.
- le paquet tm
- Cran.r: tm: Text Mining Package
- Home page
- Rdocumentation: tm
- Vignette (texte d'introduction): ntroduction to the tm Package Text Mining in R
- Extensions à tm
- tm.plugin.mail - Permet d'analyser des fichiers emails
- tm.plugin.webmining - inclut des fonctions "web scraping"
- tm.plugin.alceste - permet d'importer des fichiers au form "Aleceste" (voir aussi exemple IRaMuTeQ, un front-end graphique R gratuite qui permet de faire des analyses selon "l'école française")
Importer des fichiers avec le paquet tm
tm est le paquet "text mining" le plus populaire de R. Comme on va surtout travailler avec ce paquet, il va falloir l'installer si nécessaire.
Le paquet tm est conçu pour marcher avec une variété de formats: textes simples, articles/papiers en PDF ou Word, documents Web (HTML, XML, SGML), etc. Il fournit entre autre les fonctionnalités suivantes:
- Un dispositif pour analyser des corpus (une structure de données avec des fonctions de construction)
- Une fonction pour appliquer des fonctions à l'ensemble des textes d'un corpus.
- Des fonctions nouvelles pour l'analyse de textes
Pour utiliser tm:
library(tm)
Si nécessaire, installez ce paquet avec R-Studio ou tapez:
install.packages("tm")
Importation de documents et corpus
Le Corpus est la structure R qui représente une collection de documents que l'on veut analyser. Cette structure doit être construite en important des textes.
Il existe 2 versions:
VCorpusalias >code>Corpus. Ce type de corpus est volatile, lorsqu'on arrête R, il faut reconstruire le corpus ...PCorpus(persistant, définit un endroit unique où le corpus est stocké)
- Syntaxe de Vcorpus
- On doit indiquer le source du corpous (où le trouver) et la méthode pour lire les divers fichiers (ou autre sources).
Vcorpus(objet_source, méthode_pour_lire)- L'exemple suivant lit une source en mode "simple" et en français:
VCorpus( DirSource(txt, encoding = "UTF-8"), readerControl = list(reader=readPlain, language="fr") )
- Exemple qui utilise un reader par défaut.
VCorpus( DirSource(txt, encoding = "UTF-8")# on utilise les défauts
Voici quelques éléments à savoir lorsqu'on construit un corpus
- Identifier un type de 'source de données: soit
DataframeSource,DirSource,URISource,vectorSourceouXMLSource - Definir le reader, c-a-d. on peut définir la manière dont le texte est lu avec
readerControl=. On peut notamment définir le type de document à lire (readDOC,readPDF,readPlain,readXML,readTabular), la languelanguage=....
Le type de sources et de readers de votre installation R peut être affiché avec les commandes suivantes:
getReaders() # affiche toute la liste de readers
getSources() # affiche la liste des types de sources
Chaque source possède un lecteur (reader) par défaut. Par exemple pour DirSource, c'est readPlain.
Exemple lecture de fichiers textes du tutoriel officiel en format UTF-8
Ces fichiers se trouvent déjà dans l'installation de R, d'où l'utilisation de system.file
# Utilise le sous-répertoire tm/texts/txt dans votre bibliothèque système et assigner cette source à la variable ''txt''
txt <- system.file("texts", "txt", package = "tm")
# Lire le texte de chaque fichier et mettre dans le corpus assigné à la variable ''ovid''
ovid <- VCorpus(DirSource(txt, encoding = "UTF-8"), readerControl = list(language = "lat"))
Inspection et utilisation de corpus
Pour vérifier les contenus on peut afficher le tout ou encore juste quelques éléments, par exemple:
# print a short overview
print(ovid)
# show all
inspect(ovid)
ovid
# display the second document
ovid[[2]]
# afficher tous les textes
names(ovid)
Structures de données
Il est toujours utile de comprendre et de vérifier les types et classes de données. Un Corpus a comme type "list" > typeof(ovid) [1] "list"
Le classe d'un objet Corpus est VCorpus (alias Corpus) et qui sont des classes de type "S3".
> class(ovid)
[1] "VCorpus" "Corpus"
La classe Corpus définit une structure et un certain nombre de méthodes.
La structure est composé de:
- une liste de jeux de documents []
- une liste de documents individuels [[]]
- des métadonnées:
meta
Lecture de fichiers locaux
Pour lire un ensemble de fichier locaux,on conseille de les mettre dans un sous-répertoire d'un répertoire pour le text mining.
le "working directory" définit le répertoire de travail par défaut, selon la logique de votre plateforme. On peut modifier ce working directory.
# Afficher le répertoire de travail courrant
getwd()
[1] "C:/Users/dschneid/Documents"
# changer le répertoire de travail
setwd "C:/dks/R"
On peut utiliser la fonction file.path pour indiquer ou trouver des fichiers d'une façon indépendante de la plateforme. Si sous Windows, les fichiers se trouvent dans D:\data\exemples\txt, utilisez du code comme chemin <- file.path("d:", "data", "exemples", "txt") . Pour indiquer un sous-répertoire du répertoire courant, utilise qc. comme file.path (".", "textes").
Enfin, on peut aussi utiliser une syntaxe symple de type "Unix": D:/data/exemples/txt. Les "/" marchent sous Unix, Mac et Windows, les "\" ne marcheront pas tels quels.
Exemple: Lire plusieurs fichiers "text" dans un corpus
Voici un exemple plus concret pour Unix. Quelques documents, c-a-d. des fichiers *.text/txt simples, se trouvent dans un répertoire /home/schneide/schneide/methodo/R/ex. On va les importer dans un corpus "tm".
On vérifie le working directory et on charge la bibliothèque tm
getwd()
[1] "/home/schneide"
library(tm)
On définit le chemin:
archive_test <- file.path ("schneide", "methodo", "R", "ex")
On affiche les fichiers du répertoire:
dir (archive_test)
[1] "how-to-get-a-phd.text"
[2] "In-the-beginning-was-the-command-line-Stephenson.txt"
[3] "logic-and-qual-res.text"
...
On crée un corpus
library(tm)
corpus.source <- Corpus(DirSource(archive_test))
On examine le corpus
# short summary information
show (corpus.source)
print (corpus.source)
<<VCorpus (documents: 6, metadata (corpus/indexed): 0/0)>>
On peut aussi examiner des détails
#Affiche tout (inutile, car trop)
inspect(corpus.source)
# Afficher le premier document,
inspect(corpus.source[1])
Lire du PDF et du WORD
Il existe des filtres (readers) pour ces 2 types de documents (pas testés). A tester: il est possible qu'il vaut mieux enregistrer Word comme PDF et ensuite utiliser le filtre PDF.
Installation de xpdf
Sous Windows:
- Téléchargez l'archive zip de sourceforge (download)
- Dézippez et copiez soit les fichiers 32bit, soit les 64 bit dans un répertoire. Je conseille
c:\soft\xpdf. Donc après l'opération vous devriez y voir 9 fichiers, dont pdftotext.exe. - Editez le chemin d'exécutables de Windows et ajoutez ce répertoire. Lire Fichier_de_commande si vous ne savez pas le faire.
- Finalement, dans notre installation il manquait jpeg8.dll. On a copié/collé un fichier trouvé dans l'installation de calibre dans ce répertoire ....
Résumé de la syntaxe:
chemin <- file.path ("...")
corp <- Corpus (DirSource (chemin), readerControl=list (reader=readDOC))
corp <- Corpus (DirSource (chemin), readerControl=list (reader=readPDF))
Exemple:
library(tm)
# CHANGEZ ICI si nécessaire
# setwd ("./R")
# setwd("/home/schneide/schneide/methodo/R")
setwd("s:/methodo/R")
getwd()
archive_test <- file.path (".", "PDF")
dir (archive_test)
corpus.source_de_PDF <- Corpus(DirSource(archive_test), readerControl=list (reader=readPDF))
show (corpus.source_de_PDF)
# A la fin de ces fichiers il y a un form feed (ctrl-L)
# qu'on devrait éliminer. \r représente ce caractère
for (j in seq (corpus.source_de_PDF)) {
corpus.source_de_PDF[[j]] <- gsub ("\r","", corpus.source_de_PDF[[j]])
}
inspect (corpus.source_de_PDF[1])
# On écrit les fichiers txt dans un répertoire. Facilite la vérification.
writeCorpus(corpus.source_de_PDF, path="./PDF_txt")
Aspirer des pages web
Il existe plusieurs méthodes et paquets pour lire des pages web. Selon le type de de besoin on peut travailler avec les fonctions R "standardes" ou avec des bibliothèques ou avec une combinaison des deux. Une fois qu'on a constitué un corpus, il faut "nettoyer" les textes, voir le chapitre suivant. Ceci dit, certains paquets permettent d'effectuer des filtrages pendant la lecture.
- La fonction Corpus peut directement lire des pages sur Internet. Le résultat sera "brut" et nécessitera un traitement
- Le paquet tm.plugin.webmining est spécialisé pour aspirer des informations qui viennent de sites spécialisés (comme Reuteurs, Yahoo, etc.)
- Le paquet XML permet d'extraire des contenus selon certaines méthodes DOM.
Création d'un corpus avec tm
Les fonctions "Corpus" du paquet tm permettent aussi de lire des URLs.
Principe:
- On définit une liste d'URLS et on la donne comme paramètre à la fonction Corpus. Autrement dit une liste d'URLs est une source comme un répertoire.
liste_URLs <- c("http://tecfa.unige.ch", "http://unige.ch/")
#On construit le corpus
Corpus.brut <- Corpus(URISource(list_URLs), readerControl = list(language="fr"))
Voici un exemple un peu plus élaboré qui montre comment créer un corpus avec des pages Mediawiki. Il nous semble qu'il est préférable d'utiliser la méthode expliquée plus loin et qui profite de l'API Mediawiki. L'utilisation d'URLs avec des "blancs" ne semble pas marcher sous Linux, OK sous Windows. Il faudrait substituer des caractères, c-a-d produire un semblant d'un URL correctement "urlencoded".
library(tm)
#Une longue procédure pour avoir une liste d'articles dans un string
wiki <- "http://edutechwiki.unige.ch/fr/"
titles <- c("Activate", "Cité romaine", "Citéjob-négo", "Darfur is dying",
"Eonautes", "FacteurAcademy", "Happy Night", "InfinITy",
"J'apprends J'entreprends", "Mon entretien d'embauche",
"Timeout", "Tree Frog")
# un vecteur qui contient 12 strings vides ("")
article_list <- character(length(titles))
# on remplace par les URLs ci-dessus
for (i in 1:length(titles)) {
article_list[i] <- (paste (wiki,titles[i], sep=""))
}
#Vérification
article_list
#On construit le corpus
# wiki.source <- URISource(article_list)
wiki.source <- Corpus(URISource(article_list), readerControl = list(language="fr"))
# Vérification
wiki.source
#Inspecter le corpus
inspect(VCorpus(wiki.source))
Utilisation du paquet tm.plugin.webmining
Le paquet tm.plugin.webmining combine l'aspiration avec des filtres HTML/XML et des fonctions toutes prêtes pour analyses des contenus qui viennent de Reuters, Yahoo, etc. L'installation de ce paquet n'est pas forcément facile.
- Installation sous Windows
- Ce package nécessite Java, probablement une version pour développeurs. Ce plugin nécessite également les paquets R "XML" et "CURL" et qui seront installés automatiquement si tout va bien.
- Sous Win8, le package s'installe sans problème, mais ne trouve pas Java (Win8). Il faut soit savoir bricoler les chemins d'environnement soit (plus facile) installer Java JDK, donc le kit de développement. Ce dernier est disponible qq. part chez Oracle. Le plus facile et de chercher "Download Java JDK SE" et si vous tombez sur une page chez Oracle de type "Java SE Development Kit 8 Downloads" c'est bon ....
- Sous Linux
Tester si vous avez un compilateur Java, tapez:
- javac
Installer Java SDK de Oracle:
sudo add-apt-repository ppa:webupd8team/java
sudo apt-get update
sudo apt-get install oracle-java7-installer
Dans une console Linux (sous root), pour indiquer l'emplacement de Java, taper:
sudo R CMD javareconf
ou
sudo R CMD javareconf -e
Il faut également installer préalablement des bibliothèques de développement Curl et XML, sinon vous allez recevoir des messages d'erreurs comme ERROR: configuration failed for package ‘RCurl’, rm: cannot remove 'a.out.dSYM': Is a directory
sudo apt-get install libcurl4-openssl-devsudo apt-get install libxml2-dev
Ensuite, on peut installer le paquet. Alternativement, utilisez la facilité "Install" de RStudio.
install.packages("tm.plugin.webmining")
- Alternatives pour le web scraping
Il existe d'autres logiciels de web scraping. Trois sont listés dans la vignette R Short Introduction to tm.plugin.webmining. Il faut envisager d'utiliser ces outils externes lorsque vous téléchargez des gros volumes. Dans ce cas, on enregistre les pages dans des répertoires sur votre disque dur et on les importe dans R comme source locale.
- Charger la bibliothèque
library("tm.plugin.webmining")
On peut assez facilement enregistrer une page web. La solution pour le débutant est un simple "save as". Mais on conseille d'abord d'afficher une version "print" si elle existe (élimine déjà un certain nombre d'informations inutiles).
- Filtrage de sections inutiles
La plupart des pages web contiennent de l'information inutile
Le package tm.plugin-webmining contient un certain nombre de fonctions utiles, et notamment:
extractContentDOM (url, threshold, asText ...)
- url définit la source, par exemple une URL
- theshold définit une sorte de "force" de filtrage de div's inutiles
- asText est une variable booléenne, mettre à FALSE pour un URL
Examples:
# threshold pour une page classique
test1 <- extractContentDOM("http://tecfa.unige.ch",0.1,FALSE)
# threshold qui marche pour edutechwiki
test2 <- extractContentDOM("http://edutechwiki.unige.ch/fr/Happy_Night",0.5,FALSE)
# identique à test2
test3 <- extractContentDOM("http://edutechwiki.unige.ch/fmediawiki/index.php?title=Happy_Night&printable=yes",0.5,FALSE)
Avec la bibliothèque XML
Selon les explications de Reading HTML pages in R for text processing, par Luis, 2011
htmlTreeParse() permet de lire une page HTML et génère une structure R qui représente un arbre XML/HTML. Cette fonction connait des paramètres nombreux et fait des choses assez différentes selon la combinaison de paramètres. Notez que d'autres variantes comme la fonction xmlTreeParse existent. Elle diffèrent par rapport aux paramètres par défaut.
Une fois qu'on a "digéré" un document XML ou HTML en R, on peut ensuite utiliser tout l'éventail des technologies XML, par exemple extraire des listes de paragraphes basés sur des noms de balise.
library(XML)
doc = htmlTreeParse("http://tecfa.unige.ch/", useInternal=TRUE)
# Extraction des contenus des balises p, li, h1 et td
doc2 = unlist(xpathApply(doc, "//p|//li|//h1|//td", xmlValue))
# Virer les retour de lignes et tabs \n\t
doc3 = gsub('\\n', ' ', doc2)
doc3 = gsub('\\t', ' ', doc3)
# Joindre tout dans un seul string
doc.text = paste(doc3, collapse = ' ')
Méthode "XML" via l'API pour Mediawiki
Les MediaWiki ont une API (Application programmer interface) qui autorise l'extraction de contenus de pages sans les textes qui font partie des menus. Un bon exemple est ce wiki. On peut afficher l'API de n'importe quel MediaWiki (à moins qu'il soit vérouillé):
- http://edutechwiki.unige.ch/fmediawiki/api.php
- http://fr.wikipedia.org/w/api.php
- http://en.wikipedia.org/w/api.php
La page affiché montre quel type d'informations on peut exporter et de quelle façon. Il existe aussi une documentation complète des query sur Mediawiki.org. Nous allons d'abord nous intéresser à l'extraction du contenu d'une page sous format XML. Il va aussi falloir la nettoyer, mais moins qu'avec les méthodes qui lisent toute la page ou qui se basent sur des algorithmes de détection du corps. Voici un exemple d'une page récupéré via l'API. Si votre navigateur affiche le contenu, vous verrez que tout le contenu se trouve dans une balise "text".
Extraction du contenu d'une liste de pages
# ----------- Les bibliothèques
library(tm)
library(tm.plugin.webmining)
library(SnowballC)
library(XML)
# Path
# Linux
setwd ("~/schneide/methodo/R")
# Windows
# setwd("s:/methodo/R")
getwd()
# ----------- Mettre des pages wiki dans un corpus ------------
#Une longue procédure pour avoir une liste d'articles dans un string
# ATTENION aux noms des pages, utilisez des URL (URlEncoded)
# certains caractères ok pour le web ne passent pas en R, par exemple les "curly" apostrophes
url_start <- "http://edutechwiki.unige.ch/fmediawiki/api.php?action=parse&page="
url_end <- "&format=xml"
titles <- c("1066", "Activate", "Alice", "Argument_Wars", "CeeBot_4", "Chevron", "Cité_romaine", "Citéjob-négo", "Cyberbudget", "Darfur_is_dying", "E-psych", "Elude", "Energy_City", "Envers_et_contre_tout", "Eonautes", "FacteurAcademy", "Foodforce", "Get_the_glass", "Glucifer", "Halte_aux_catastrophes", "Happy_Night", "I-progress", "ICE-D", "InfinITy", "Ivy%E2%80%99s_Meadow", "J%27apprends_J%27entreprends", "K-ROBOT", "Mon_entretien_d%27embauche", "MySQLgame", "Oiligarchy", "Orbitrunner", "Petits_Détectives", "Phun", "Play_the_news", "Real_Lives", "RobotProg", "ScienceMuseum", "September_12th", "StarBankTheGame", "Super_Kimy", "SuperBetter", "TechnoCity", "The_Great_Flu", "The_Traveler_IQ_Challenge", "Timeout", "Tree_Frog", "Typershark", "Une_journée_au_fil_de_l%27eau")
# un vecteur qui contient 12 strings vides ("")
article_list <- character(length(titles))
# on remplace par les URLs ci-dessus
for (i in 1:length(titles)) {
article_list[i] <- (paste (url_start,titles[i],url_end, sep=""))
}
#Vérification
article_list
# On construit le corpus en utilisant un reader fait par nous et
# Cette fonction extrait juste l'élément XML "text" (voir l'API des mediawiki)
readMWXML <-
readXML (spec = list (content = list ("node", "//text"),
heading = list ("attribute", "//parse/@title")
),
doc=PlainTextDocument())
# Attention: Casse (notamment sous Ubuntu) si les URLs ne sont pas correctement encodés
wiki.source <- VCorpus(URISource(article_list, encoding="UTF-8"),
readerControl=list(reader=readMWXML, language="fr"))
# On change les "id" (titres à la place d'URLs illisibles)
for (j in seq.int (wiki.source)) {
meta(wiki.source[[j]],"id") <- titles[j]
}
# Ajouter une balise html autour du tout - c'est du bon vodoo
wiki.source <- tm_map (wiki.source, encloseHTML)
# Ecrire les fragments HTML dans des fichiers (inutile pour une analyse statistique, mais permet l'inspection)
writeCorpus(wiki.source, path="./wiki_txt_html")
On a maintenant une série de documents "bruts" qui incluent le "corps" d'une page wiki et qu'il va falloir nettoyer. Voici le début d'un fichier
<div style="border:1px solid grey;background-color:#FFAABB;padding:7px; margin-bottom: 10px;">
<p style="text-align:center;font-weight:bold;">
Page réalisée dans le cadre du cours <a rel="nofollow" class="external text" href="http://vip-tetris.mixxt.com/">Jeux Vidéo Pédagogiques (VIP)</a><br /> (volée "Tetris" 2013-2014) de la formation <a rel="nofollow" class="external text" href="http://tecfalabs.unige.ch/maltt/master/qui-sommes-nous/">maltt</a>, au <a rel="nofollow" class="external text" href="http://tecfa.unige.ch/">TECFA</a>.</p>
<p style="text-align:center;font-weight:bold;">
Cette page est une ébauche en cours de réalisation. Ne pas citer.</p>
</div>
.....
Extraction des pages d'une catégorie wiki
L'API d'un Médiawiki permet également de lister les membres d'une catégorie. La documentation API:Categorymembers explique comment. Si vous pensez analyser tous les articles d'une catégorie, voilà une solution plus simple pour extraire une liste d'articles dans un Médiawiki.
Pour extraire une liste de noms, on peut utiliser l'URL suivant:
Explications:
action=queryindique qu'on transmet une requêtelist=categorymembersindique qu'on veut lister les membres d'une categoriecmtitle=Category:Jeux_p%C3%A9dagogiquesimportant: Définit le nom de la catégorie (copié/collé depuis l'URL de la page, http://edutechwiki.unige.ch/fr/Cat%C3%A9gorie:Jeux_p%C3%A9dagogiques)cmtype=pageindique qu'on en prend que les pages (et pas les sous-catégories)format=xmlindique le format de sortie (voir API:Data formats)
Le fichier XML que le serveur wiki produit ressemble à cela:
<api><query>
<categorymembers>
<cm pageid="3731" ns="0" title="1066" />
<cm pageid="6542" ns="0" title="3D World Farmer" />
<cm pageid="6541" ns="0" title="3D WorldFarmer" />
......
</categorymembers>
Voici le code:
# ----------- Les bibliothèques
library(tm)
library(tm.plugin.webmining)
library(SnowballC)
library(XML)
library(RCurl)
library(httr)
# Path
# Linux
setwd ("~/schneide/methodo/R")
# Windows
# setwd("s:/methodo/R")
getwd()
# Extraire toutes les pages d'une catégorie,
# InternalNodes=TRUE est du Vodoo crucial à ajouter.
categ <- "http://edutechwiki.unige.ch/fmediawiki/api.php?action=query&list=categorymembers&cmtitle=Category:Jeux_p%C3%A9dagogiques&cmlimit=500&cmtype=page&format=xml"
liste_XML <- xmlTreeParse(categ,useInternalNodes = TRUE)
#vérifier
class (liste_XML)
# extraire les éléments qui définissent une page, sans les pages qui ont 'Template' dans leur titre
# Xpath qui trouve les mauvais: //cm[contains(@title,'Template')]
liste_XML2 <- xpathSApply(liste_XML, "//cm[not(contains(@title,'Template'))]")
# on produite deux vecteurs avec titres et ID wiki de la page
liste_titres = sapply(liste_XML2, function(el) xmlGetAttr(el, "title"))
liste_ids = sapply(liste_XML2, function(el) xmlGetAttr(el, "pageid"))
# vérifier
liste_titres
liste_ids
# ----------- Mettre des pages wiki dans un corpus ------------
# début et fin de l'URL. Notez le "pageid" qui va nous sortir un article avec sa "pageid"
url_start <- "http://edutechwiki.unige.ch/fmediawiki/api.php?action=parse&pageid="
url_end <- "&format=xml"
article_id_list <- character(length(liste_ids))
for (i in 1:length(liste_ids)) {
article_id_list[i] <- (paste (url_start,liste_ids[i],url_end, sep=""))
}
#Vérification
article_id_list
# On construit le corpus en utilisant un reader fait par nous et
# Cette fonction extrait juste l'élément XML "text" (voir l'API des mediawiki)
readMWXML <-
readXML (spec = list (content = list ("node", "//text"),
heading = list ("attribute", "//parse/@title")
),
doc=PlainTextDocument())
# On télécharge, prend qqs. dizaines de secondes
wiki.source <- VCorpus(URISource(article_id_list, encoding="UTF-8"),
readerControl=list(reader=readMWXML, language="fr"))
# On change les "id" (titres à la place d'URLs illisibles)
for (j in seq.int (wiki.source)) {
meta(wiki.source[[j]],"id") <- liste_titres[j]
}
# Ajouter une balise html autour du tout - c'est du bon vodoo
wiki.source <- tm_map (wiki.source, encloseHTML)
# Afficher / vérifier la liste de documents une dernière fois...
names(wiki.source)
# Ecrire les fragments HTML dans des fichiers (inutile, mais permet l'inspection)
writeCorpus(wiki.source, path="./wiki_txt_html")
Problèmes
- Error
- 1: Extra content at the end of the document
- Si vous avez le message Error: 1: Extra content at the end of the document, il est probable que R demande un faux URL au wiki. Celui-ce répond avec un message d'erreur.
- Solution: Utiliser des noms des URLs qui sont strictement URL-encoded (donc pour des pages wiki, il faut par exemple remplacer les " " par des "_".
Vous pouvez utiliser le debugger pour trouver un URL cassé (pas trouvé d'autre moyen):
- Dans R-Studio: Menu Debug; cocher "On Error -> Break in code"
- Dans le panneau Traceback, sélectionner la ligner readerControl$....
- Dans le panneau Values, ouvrir elem, puis regarder uri
On va réutiliser ces données dans le chapitre suivant.
Transformations de Corpus "tm"
Il existe un certain nombre de fonctions qui permet de "nettoyer" le texte d'un Corpus. La construction <tm_map> permet de appliquer une transformation à l'ensemble des textes d'un corpus. Parfois les fonctions fournies par "tm" ne suffisent pas et on doit faire des calculs plus "manuels".
Les fonctions de transformation
Partant avec un corpus, appelé corpus0, on peut effectuer plusieurs opérations de transformation, par exemple:
- stripWhitespace()
- Enlève les blancs en trop
corpus1 <- tm_map(corpus0, stripWhitespace)
- tolower();
- Met tous les mots en minuscules
corpus2 <- tm_map(corpus1, tolower)
- removeWords(....),
- Enlever des mots
- Par exemple des stopwords en Anglais:
corpus3 <- tm_map(corpus2, removeWords, stopwords("english")) - ou en français:
corpus5 <- tm_map(corpus3, removeWords, stopwords("french")) - ou une liste explicite de mots:
xxx = tm_map(corpus2, removeWords, c("Daniel", "Kaspar", "TECFA"))
- removePunctuation()
- Enlever les ponctuations
- removeNumber()
- Enlever des nombres
- stemDocument()
- Faire du stemming
library(SnowballC)
corpus9 = stemDocument(corpusx, language = meta(corpusx, "language"))
On peut obtenir cette liste des transformations de tm avec la fonction:
getTransformations()
Certaines manipulations doivent se faire avec d'autres paquets ou encore avec les fonctions de base de R. Notamment la substitution de caractères:
- Remplacer des caractères (merci à Graham Williams)
for (j in seq (corpus0)) {
corpus0[[j]] <- gsub ("/", " ", corpus0[[j]])
corpus0[[j]] <- gsub ("@", " ", corpus0[[j]])
......
}
On peut aussi utiliser des expression régulières, par exemple une qui tue une balise XML/HTML dans un string.
gsub("<.*/>","",string)
Ceci dit, les regexp ne marchent pas très bien pour enlever des balises HTML. Il vaut mieux utiliser une fonction du plugin tm.plugin.webmining
wiki.clean <- tm_map (wiki.source, extractHTMLStrip, encoding="UTF-8")
Exemple EduTechWiki (suite)
L'exemple suivant transforme les textes d'un corpus qu'on a crée ci-dessus avec la méthode API/XML, à relire dans la section Extraction des pages d'une catégorie wiki
# ------------------------------- Nettoyage du texte
# Mettre tout en minuscules
# Le code suivant ne semble PAS marcher !!!
# wiki.clean3 <- tm_map (wiki.clean2c, tolower)
# Utiliser cela
wiki.cl1 <- tm_map(wiki.source, content_transformer(tolower))
# Tuer les balises. Attention à l'encodage !!
# Empêche le stemmer de marcher :(
# wiki.cl2 <- tm_map (wiki.cl1, extractHTMLStrip, encoding="UTF-8")
# Ceci marche mieux
wiki.cl2 <- tm_map(wiki.cl1, content_transformer(extractHTMLStrip))
# kill_chars est une fonction pour nettoyage custom
# ... ne pas utiliser une boucle "for" pour un Corpus, ou il est naze
# curly quotes = \u2019
(kill_chars <- content_transformer (function(x, pattern) gsub(pattern, " ", x)))
tm_map (wiki.cl2, kill_chars, "\u2019")
tm_map (wiki.cl2, kill_chars,"'")
tm_map (wiki.cl2, kill_chars,"[«»”“\"]")
tm_map (wiki.cl2, kill_chars,"\\[modifier\\]")
# enlever les ponctuations qui restent
wiki.cl3 <- tm_map (wiki.cl2, removePunctuation, preserve_intra_word_dashes = TRUE)
# Tuer les mots fréquents (stop words)
wiki.essence <- tm_map (wiki.cl3, removeWords, stopwords("french"))
# Extraire les racines. La bibliothèque SnowballC doit être installée.
# Ne semble pas marcher après des transformations fait sans "tm_map" !!
getStemLanguages()
wiki.racines <- tm_map (wiki.essence, stemDocument, language="french")
# Enlever des blancs s'il en reste
wiki.racines <- tm_map (wiki.racines, stripWhitespace)
# test
wiki.racines[[2]]
class(wiki.racines)
A ne pas faire:
- utiliser une boucle pour remplacer des valeurs dans la matrice
- Utilise toLower tel quel (voir le code ci-dessus)
Le stemmer ne marchera plus ....cela m'a fait perdre une journée ou plus !!
# Replacing curly quotes does not work because it can't distinguish from straight quotes)
# Such behavior is not acceptable whatever the programmer's reasons could be.
# Anyhow, if you use some UTF-8 hex code it may work.
# U+2019 = ’
# \0xE2\0x80\0x98
for (j in seq.int (wiki.clean2a)) {
wiki.clean2a[[j]] <- gsub("\u2019"," ",wiki.clean2a[[j]])
}
for (j in seq.int (wiki.clean2a)) {
wiki.clean2a[[j]] <- gsub("'"," ",wiki.clean2a[[j]])
}
for (j in seq.int (wiki.clean2a)) {
wiki.clean2a[[j]] <- gsub("[«»”“\"]"," ",wiki.clean2a[[j]])
wiki.clean2a[[j]] <- gsub("\\[modifier\\]"," ",wiki.clean2a[[j]])
}
Matrices documents-termes et analyses de fréquences
Note: on n'utilise pas forcément les mêmes données dans les exemples suivant. La majorité des cas utilisent une liste "normale" de mots, mais sans les "stop word"
Créer des matrices
Une matrice documents-termes (Angl: Document Term Matrix (DTM) liste la fréquence de mots par document. Il existe deux variantes, un matrice "documents par termes" ou une matrice "termes par documents" comme c'est expliqué dans l'article text mining.
Pour construire une matrice il faut utiliser soit DocumentTermMatrix, soit TermDocumentMatrix. Le corpus doit inclure les meta data qui on pu disparaître lors d'opérations de transformation, donc il faut les remettre comme ci-dessous
# Du Vodoo pour de nouveau créer un "vrai corpus"
wiki.mots <- Corpus(VectorSource(wiki.racines))
matrice_docs_termes <- DocumentTermMatrix(wiki.mots)
Reduction de la matrice
Notre matrice_termes_docs contient environ 2400 mots et que l'on pourra réduire
inspect(matrice_termes_docs)
removeSparseTerms enlève les mots que l'on retrouve dans moins de 40 et 60% des documents. Autrement dit, plus on réduit la proportion, moins on des mots.
inspect(removeSparseTerms(matrice_termes_docs, 0.4))
inspect(removeSparseTerms(matrice_termes_docs, 0.6))
Voilà les mots aimés par tous les auteurs ....
inspect(removeSparseTerms(matrice_termes_docs, 0.01))
<<TermDocumentMatrix (terms: 32, documents: 12)>>
Non-/sparse entries: 384/0
Sparsity : 0%
Maximal term length: 12
Weighting : term frequency (tf)
Docs
Terms 1 2 3 4 5 6 7 8 9 10 11 12
20132014 1 1 1 1 1 1 1 1 1 1 1 1
cadre 1 1 1 1 4 1 1 1 1 6 3 1
citer 1 1 1 1 1 1 1 2 1 1 1 1
contenu 5 10 2 7 6 3 6 7 4 6 5 2
cours 4 3 2 2 4 3 4 5 2 5 2 2
description 2 2 2 2 2 2 3 2 2 5 3 2
doit 5 6 2 1 16 2 8 8 2 5 10 9
ébauche 1 1 1 1 1 1 1 1 1 1 1 1
enseigné 4 8 2 4 6 2 5 5 3 2 5 2
fait 3 8 1 1 1 1 3 1 2 4 3 2
formation 1 1 1 1 1 3 1 1 1 1 1 1
forts 4 3 2 2 4 2 4 4 3 4 4 4
intégration 2 3 2 3 4 2 4 5 2 3 3 3
jeu 37 38 14 20 26 8 46 50 33 69 29 14
jeux 4 11 1 3 3 1 5 3 1 4 1 2
joueur 33 15 1 2 35 2 22 25 13 23 1 22
logiciels 2 2 2 2 2 2 3 4 4 2 2 4
maltt 1 1 1 1 1 1 1 1 1 1 1 1
page 2 2 2 2 2 2 4 2 2 4 2 2
pédagogiques 3 5 3 3 3 3 4 3 3 5 3 4
point 2 1 2 3 2 2 2 1 6 7 5 5
points 6 6 2 3 6 2 9 6 5 12 4 6
principes 2 2 2 3 3 2 2 2 3 3 3 3
réalisation 1 1 1 1 1 1 1 1 1 1 1 1
réalisée 1 1 1 1 1 1 1 1 1 1 1 1
similaires 2 2 2 2 3 2 2 2 2 2 2 2
sommaire 1 1 1 1 1 1 1 1 1 1 1 1
tecfa 1 1 1 1 1 1 1 1 1 1 1 1
tetris 1 1 1 1 1 1 1 1 1 1 1 1
vidéo 1 1 1 1 1 1 2 3 1 3 2 1
vip 1 1 1 1 3 1 1 1 1 1 1 1
volée 1 1 1 1 1 1 1 1 1 1 1 1
Voici une liste (tronquée) de mots que l'on retrouve dans au moins 40% des documents:
Non-/sparse entries: 848/160
Sparsity : 16%
Maximal term length: 13
Weighting : term frequency (tf)
Docs
Terms 1 2 3 4 5 6 7 8 9 10 11 12
20132014 1 1 1 1 1 1 1 1 1 1 1 1
accès 2 2 0 3 8 1 4 2 1 1 2 0
actions 1 1 0 1 3 0 3 0 1 1 3 0
agit 5 0 1 1 0 1 3 0 2 2 1 4
ainsi 4 0 1 0 2 0 5 3 0 4 1 1
apprentissage 6 8 0 0 11 0 1 4 1 4 4 5
aussi 6 2 1 1 0 1 0 3 1 5 3 1
avant 1 3 1 0 1 0 1 1 0 3 1 0
bien 3 6 0 1 5 3 6 1 0 5 2 0
but 6 1 0 0 2 0 4 1 1 3 1 3
.......
jeu 37 38 14 20 26 8 46 50 33 69 29 14
jeux 4 11 1 3 3 1 5 3 1 4 1 2
jouer 0 3 2 0 2 1 2 2 0 1 0 2
joueur 33 15 1 2 35 2 22 25 13 23 1 22
logiciels 2 2 2 2 2 2 3 4 4 2 2 4
long 1 2 0 1 1 0 4 1 0 1 0 1
lors 5 1 0 0 2 0 1 4 1 9 1 0
.......
vip 1 1 1 1 3 1 1 1 1 1 1 1
volée 1 1 1 1 1 1 1 1 1 1 1 1
Visualisation d'une matrice termes-documents
La matrice suivante donne la même vision que ci-dessus, mais avec un graphique. Enfin pour augmenter la lisibilité on baissé le seuil de mots à montrer à la présence d'au moins 80% des documents (0.2 = absent dans moins que 20%).
Pour créer ces visualisations, on ne peut pas utiliser les données brutes (enfin j'imagine qu'il doit bien exister une bibliothèque pour cela). A la place on fait une transformation qui nous ramène à des valeurs < 1 et qui représentent le "poids" d'un terme comme expliqué dans l'article text mining. Mais attention, on ne s'est pas documenté sur l'algorithme (weightTf) utilisé, il y a peut-être mieux à faire....
# Créer une DTM avec des poids normalisées
mtd.norm <- as.matrix(removeSparseTerms(
TermDocumentMatrix(wiki.mots, control=list(weighting=weightTf)),
0.2))
corrplot (mtd.norm, is.corr=FALSE)
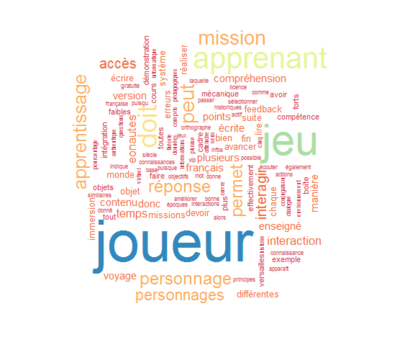
On voit que le mot "jeu" domine largement "le débat" dans tous les textes. Pour mieux faire ressortir les autres distributions, on peut soit éliminer ces mots, soit utilisé une autre matrice qui donne moins de poids au termes fréquents et ommnipreésents, soit les deux.
Ci-dessous on utilise un style de "programmation" plus dense.
- La matrice termes-documents est est transformé en matrice termes-documents "weighted" selon la méthode TfIdf expliquée dans Text mining (la fréquence du terme dans un document pondéré par la fréquence du terme dans le corpus)
- Pour finir, on l'a épuré de tous les mots qui se trouvent dans moins de 40% de documents.
# Créer une DTM avec des poids TFIdf
mtd.TfIdf <- as.matrix(
removeSparseTerms(
TermDocumentMatrix(
tm_map(wiki.mots),
control=list(weighting=weightTfIdf)
),
0.4)
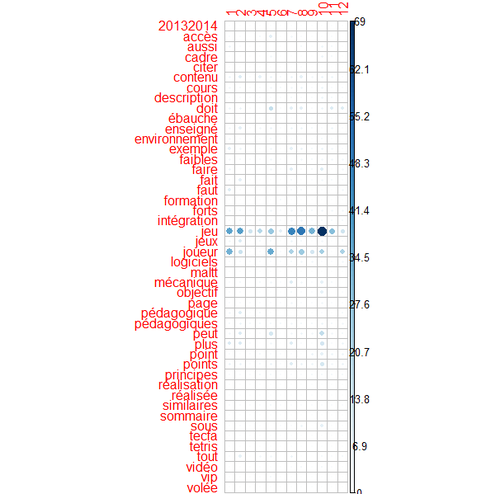
On observe que le mot "jeu" a disparu. Etant donné qu'il existait dans chaque document il n'était pas discriminant.
Ci dessous, on montre comment manuellement enlever des mots dans un jeu de données plus large. Le corpus mieux nettoyé et correctement "stemmé".
# Créer une DTM avec des poids TFIdf et sans 3 mots
mtd.norm_sans <- as.matrix(
removeSparseTerms(
TermDocumentMatrix(
tm_map(wiki.mots, removeWords, c("jeu", "jeux", "joueur")),
control=list(weighting=weightTfIdf)
),
0.2)
)
corrplot (mtd.norm_sans, is.corr=FALSE)
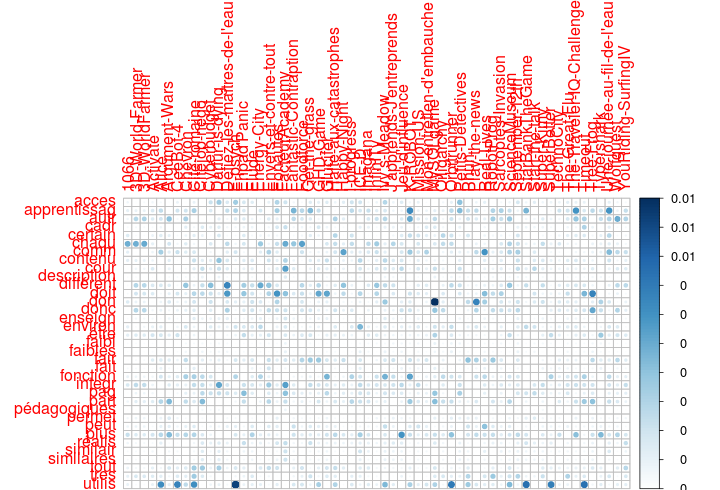
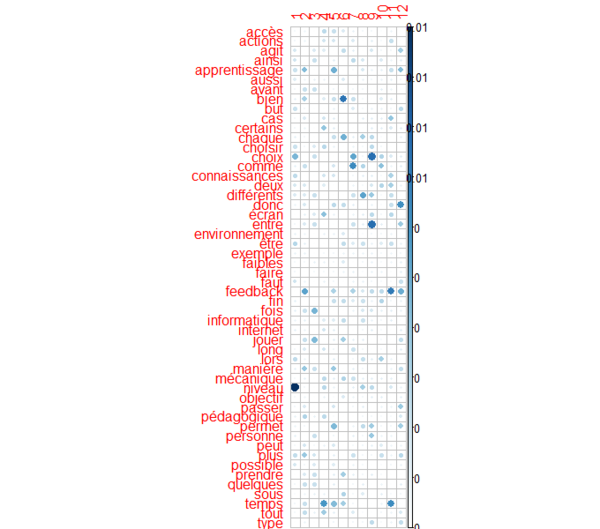
Voici encore une fois une matrice TFIdf d'un petit corpus On utilise un plus petit seuil = 20%
library(corrplot)
mtd.TfIdf2 <- as.matrix(removeSparseTerms(
TermDocumentMatrix(wiki.mots, control=list(weighting=weightTfIdf)),
0.2))
# Plot simple
corrplot (mtd.TfIdf2, is.corr=FALSE)
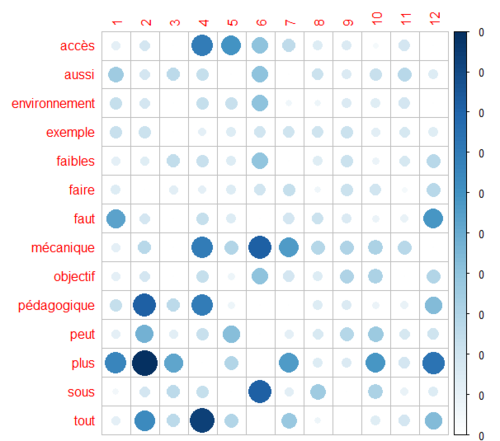
Tableaux de termes fréquents
Rappelons que nos textes on été épurés (pas de stop words, des racines). Ceci dit, il nous semble que le "stemming" ne marche pas bien (à vérifier pourquoi). Le code suivant liste tous les mots qui sont utilisé au moins 30 fois.
findFreqTerms(matrice_docs_termes, 30)
Résultat:
[1] "apprentissage" "bien" "choix" "comme"
[5] "contenu" "cours" "deux" "doit"
[9] "enseigné" "être" "exemple" "faibles"
[13] "faire" "fait" "feedback" "fin"
[17] "forts" "intégration" "jeu" "jeux"
[21] "joueur" "logiciels" "mécanique" "mission"
[25] "niveau" "pédagogiques" "permet" "peut"
[29] "plus" "point" "points" "principes"
[33] "tout" "utilisateur"
Associations mot avec un mot
Quels mots sont utilisés souvent avec "feedback" dans un document ?
# find associations with a word
findAssocs(matrice_termes_docs, "feedback", 0.8)
feedback
explication 0.84
haut 0.83
milieu 0.80
findAssocs(matrice_termes_docs, "feedback", 0.7)
feedback
explication 0.84
haut 0.83
milieu 0.80
premier 0.77
car 0.76
expérience 0.76
etc 0.71
travers 0.71
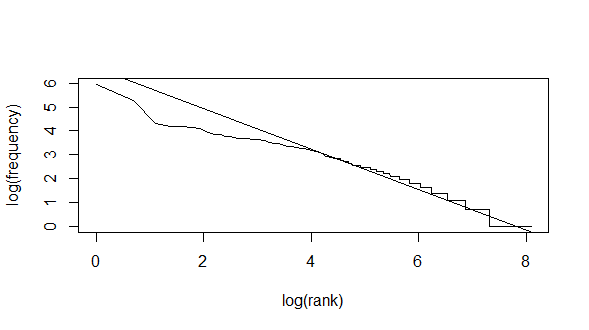
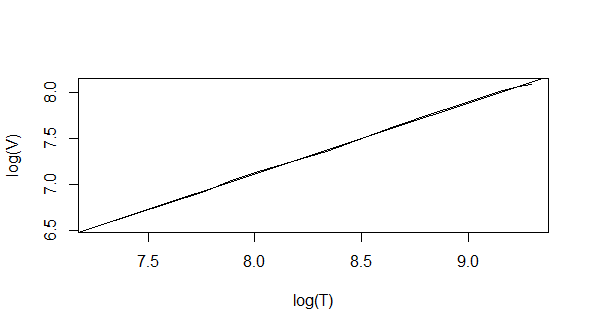
Corroboration des lois de Zipf et de Heap
Lire:
- La loi de Zipf (Wikipedia)
- Heaps Law (Wikipedia/en)
Zipf_plot(matrice_termes_docs)
(Intercept) x
6.6338162 -0.8466039
Heaps_plot(matrice_termes_docs)
(Intercept) x
0.9737307 0.7676657
Word Clouds
Dans R, il existe plusieurs méthodes pour créer des wordcloud. Le paquet wordcloud semble être populaire.
Palette de couleurs
Les words clouds peuvent utiliser des palettes de couleurs
# Afficher les palettes des couleurs pour pouvoir mieux choisir
display.brewer.all()

La bibliothèque wordcloud
La fonction wordcloud(...) de la bibliothèque wordcloud permet de créer des wordcloud en utilisant plusieurs paramètres, par exemple:
- words
- Soit un objet de type Corpus ou character vector, soit une liste
- freq
- Un vecteur de fréquences. Ce paramètre est obligatoire si words est une liste (?)
- scale=vecteur avec 2 nombres
- Définit l'empan de la taille des mots (plus grand/ plus petit)
- min.freq = nombre
- Définit un seul de fréquence. Au-dessous les mots sont éliminés
- colors = liste de couleurs
- du plus fréquent au moins fréquent
- max.words = nombre
- Max. mots à afficher. Les moins fréquents sont éliminés.
- vfont=c("nom","variante"))
- définit la fonte.
- Wordclouds faits directement à partir d'un objet de type Corpus
Ci-dessous on crée plusieurs words clouds pour des documents invidiuels. wiki.racines est un object Corpus du paquet tm.
# Wordclouds
library(wordcloud)
wordcloud(wiki.racines,
scale=c(5,0.1), rot.per=0.35,
min.freq=3, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
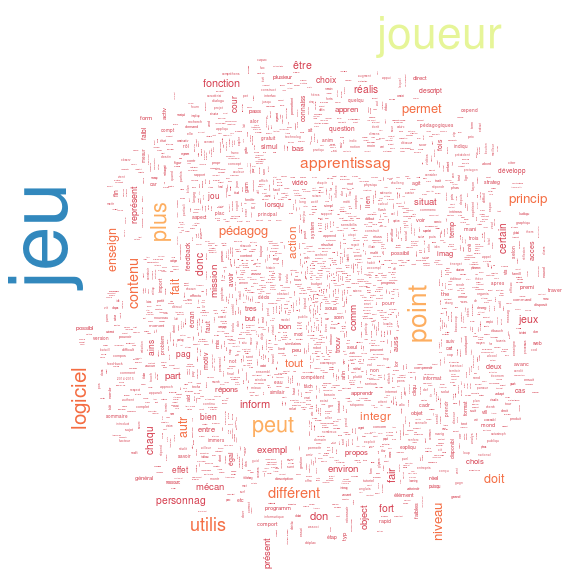
l'exemple suivant ne marchera pas car un élément du corpus est de type "TextDocument" (défini par le paquet NLP)
> class(wiki.racines[[8]])
[1] "PlainTextDocument" "TextDocument"
wordcloud(wiki.racines[[8]],
scale=c(5,0.1), rot.per=0.35,
min.freq=3, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
Pour que cela marche il faut extraire la liste des mots, par exemple avec la fonction words
wordcloud (words (wiki.racines[[2]]),
scale=c(5,0.1), rot.per=0.35,
min.freq=3, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
wordcloud(words (wiki.racines[[3]]),
scale=c(5,0.1), rot.per=0.35,
min.freq=3, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
- Comparaison word clouds de fiches (pages wikis) sur un jeu pédagogique
- Word clouds avec des matrices documents termes
Ci-dessous des clouds qui affichent des matrices documents termes d'un corpus
#Communality clouds
mtd <- as.matrix(matrice_termes_docs)
comparison.cloud(mtd,random.order=FALSE,
scale=c(5,0.5), rot.per=0.35,
max.words=50, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
commonality.cloud(mtd,random.order=FALSE,
scale=c(5,0.5), rot.per=0.35,
max.words=50, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)
Etant donne que le mot "jeu" domine trop, on pourrait l'éliminer
#Without the "jeu"
wiki.sans_jeu <- tm_map(wiki.mots, removeWords, c("jeu", "jeux"))
mtd2 <- TermDocumentMatrix(wiki.sans_jeu)
mtd2 <- as.matrix(mtd2)
commonality.cloud(mtd2,random.order=FALSE,
scale=c(2,0.5), rot.per=0.35,
max.words=50, use.r.layout=FALSE,
colors= brewer.pal(8,"Spectral")
)

Analyse typologique hiérarchique
Comme la DTM est un peu longue (plus que 6557, on va utiliser un seuil de 80% (enlever les mots que l'on retrouve dans moins de 80% des documents). Si on gardais tout la variété, les documents serait trop spécifiques en terme de distance.
# Créer une DTM avec des poids TfIdf
mtd4.TfIdf <- (DocumentTermMatrix(wiki.mots, control=list(weighting=weightTfIdf)))
# show docs X terms
dim(mtd4.TfIdf)
# Inspecter premiers 5 docs et les termes 8000-8005
inspect (mtd4.TfIdf[1:5, 500:505])
mtd4.TfIdf02 <- as.matrix(removeSparseTerms(
DocumentTermMatrix(wiki.mots, control=list(weighting=weightTfIdf)),
0.79))
dim(mtd4.TfIdf02)
# compute a distance matrix and then apply hclust
dist4 <- dist(mtd4.TfIdf, method="euclidean")
dist4
hc4 <- hclust(dist4, method="ward.D2")
plot(hc4)
# same with other params
dist5 <- dist(mtd4.TfIdf02, method="euclidean")
dist5
hc5 <- hclust(dist5, method="ward.D2")
plot(hc5)
Voici les résultats de l'analyse typologique hiérarchique (distance euclidiens, methode D2 de Ward)
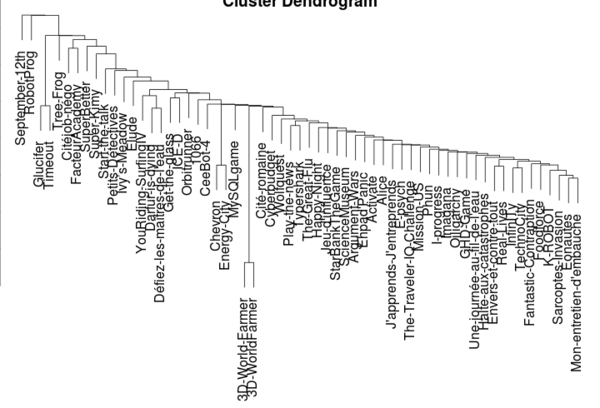
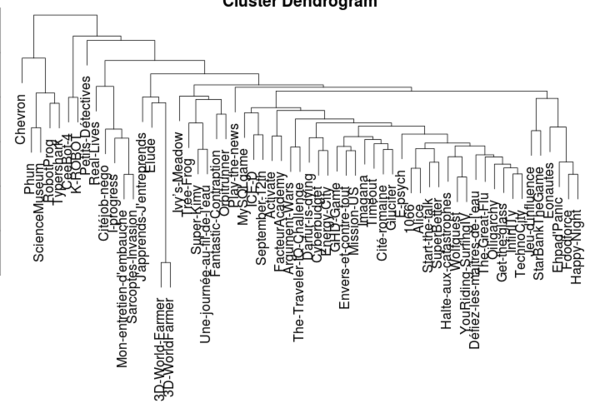
Juste pour le fun, on peut aussi produire un graphique pour la matrice à distance (fait pour la "dist5")
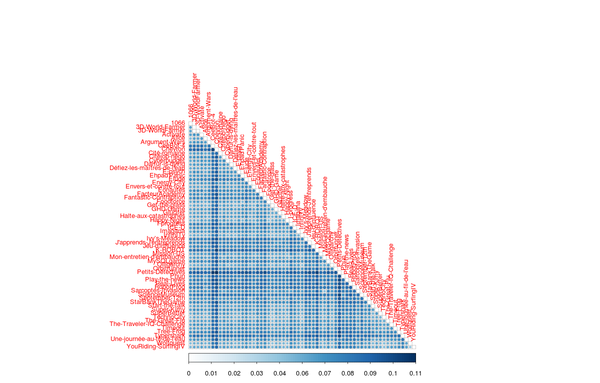
Voir aussi:
Analyse typologique avec kmeans
- ---------------------- Kmeans Cluster simple
kmeans4 <- kmeans (mtd4.TfIdf02, centers=6)
# afficher les cluster (c.f. ci-dessous)
kmeans4$cluster
# plot les cluster dans 2 dimensions principales
plotcluster (mtd4.TfIdf02, kmeans4$cluster)
# afficher l'utilisation des cluster
table (kmeans4$cluster)
Voici les résultat bruts. On voit que la plupart des pages sont du type "6"
> kmeans4$cluster
1066 3D-World-Farmer 3D-WorldFarmer
6 8 8
Activate Alice Argument-Wars
6 7 6
CeeBot-4 Chevron Cité-romaine
1 9 6
Citéjob-négo Cyberbudget Darfur-is-dying
5 6 6
Défiez-les-maîtres-de-l'eau E-psych Ehpad'Panic
6 6 3
Elude Energy-City Envers-et-contre-tout
8 6 6
Eonautes FacteurAcademy Fantastic-Contraption
3 6 2
Foodforce Get-the-glass GHD-Game
3 6 6
Glucifer Halte-aux-catastrophes Happy-Night
6 6 3
I-progress ICE-D Imagana
5 6 6
InfinITy Ivy’s-Meadow J'apprends-J'entreprends
6 2 8
Jeu-d'influence K-ROBOT Mission-US
3 1 3
Mon-entretien-d'embauche MySQLgame Oiligarchy
5 6 6
Orbitrunner Petits-Détectives Phun
2 10 7
Play-the-news Real-Lives RobotProg
7 4 7
Sarcoptes-Invasion ScienceMuseum September-12th
5 7 6
StarBankTheGame Start-the-talk Super-Kimy
3 6 2
SuperBetter TechnoCity The-Great-Flu
6 6 6
The-Traveler-IQ-Challenge Timeout Tree-Frog
5 6 2
Typershark Une-journée-au-fil-de-l'eau Wolfquest
7 6 6
YouRiding-SurfingIV
6
>table (kmeans4$cluster)
1 2 3 4 5 6 7 8 9 10
2 5 7 1 5 29 6 4 1 1
Il faudrait maintenant afficher qc. comme les 10 mot les plus populaires par type....
Composantes principales
Il existe pleins de méthodes et de paquets qui font des sortes d'analyses en composantes principales. A développer.
Kernlab permet de faire des analyses non-linéaires (enfin on a pas vérifié si la méthode par défaut est appropriée pour analyses une matrice DTM.
library (kernlab)
pca2 <- kpca (as.matrix(mtd4.TfIdf02), features=2)
plot( rotated(pca2), pty="s",
xlab="1st Principal Component", ylab="2nd Principal Component" )