Introduction à la modélisation des données avec R
Cet article est en construction: un auteur est en train de le modifier.
En principe, le ou les auteurs en question devraient bientôt présenter une meilleure version.
Introduction
Étant R un logiciel utilisé très souvent dans le contexte du Data Science, il existe des nombreuses possibilités pour modéliser des données, notamment à travers une grande variété de paquets externes. La modélisation des données est un sujet très vaste et qui incorpore différents approches philosophiques, théoriques et pratiques. Certaines sont déjà utilisées depuis longtemps en sciences sociales, comme les statistiques inférentielles de type fréquentiste, tandis que d'autres commencent à apparaître (e.g. statistiques Bayésiennes, machine learning), en dépit du fait qu'elles existent déjà depuis plusieurs années (voir siècles pour les statistiques Bayésiennes). Le point de contact parmi ses différentes approches est souvent identifié comme la séparation entre le signal et le bruit. Même si cette définition est probablement trop abstraite pour qu'elle puisse être utile sans une expérience préalable dans le domaine, elle permet d'éviter de tomber dans un champ plutôt qu'un autre. D'autres définitions qu'on peut parfois lire dans des livres de textes en sciences sociales (e.g. la réduction des données à un indicateur statistique ou encore la quantification de l'incertitude) ne sont souvent valable que dans une approche spécifique.
Dans cette page, nous proposons d'abord une description du fonctionnement technique de la modélisation en R, notamment en référence aux caractéristiques de Input et Output des fonctions qui s'occupent de modéliser des données. Ensuite, nous proposons quelques exemples d'application se référant à différentes approches que nous n'aurons sûrement pas la possibilité d'approfondir, mais dont il est peut-être utile de connaître l'existence.
Prérequis
Cet article présuppose deux types de connaissances préalables :
- Techniques, relatives à l'utilisation de R
- Théoriques, relatives à des connaissances en modélisation des données
Connaissances techniques
Cette page fait partie du parcours guide Pensée computationnelle avec R et nécessitent pourtant au moins une première exposition à l'environnement de R et à la programmation. Si vous n'avez pas suivi le parcours guidé et vous avez une expérience limité avec R ou la programmation en général, la lecture de ces pages peut être utile :
De plus, même si les exemples proposés dans la page peuvent être reproduit dans tout environnement qui supporte R, l'utilisation de RStudio est conseillée, voir à ce propos :
Connaissances théoriques
Cet article aborde des concepts statistiques qui nécessitent au moins une expérience de base au préalable avec l'analyse de données quantitatives. Si vous avez suivi un cours en méthodologie de recherche en général ou en statistiques en particulier et mené une analyse statistique (e.g. régression linéaire, t-test, ANOVA, ...), vous pouvez suivre le contenu, même si très probablement certains informations seront nouvelles.
Si vous n'avez pas d'expérience au préalable ou vous préférez revoir certains principes avant d'aborder le contenu de cette page, vous pouvez vous référez au Manuel de recherche en technologie éducative, notamment aux sections consacrées aux données et analyses quantitatives :
Les informations du manuel sont largement généralisables à tout contexte en sciences sociales, même en dehors de la technologie éducative.
Principes techniques de la modélisation des données
La modélisation des données en R, sauf quelques exceptions, suit toujours le même schéma de base dans lequel les données représentent l'Input et le modèle représente l'Output. Entre les deux, une fonction compute le modèle selon une formule (avec éventuellement d'autres paramètres) et le met à disposition, normalement avec des informations corollaires qui permettent, par exemple, d'évaluer sa pertinence ou de le visualiser graphiquement.
DONNÉES -> FONCTION(FORMULE, ....) -> MODÈLE
Modéliser à travers une formule
La plupart des paquets qui s'occupent de la modélisation des données en R utilise une syntaxe pareille, ou parfois très similaire, pour expliciter les caractéristiques du modèle à utiliser, c'est-à-dire la relation entre les données/variables qui font partie du jeu des données.
Voyons toute de suite un schéma essentiel qui sera ensuite adapté à des fonctions existantes :
1 # Exemple de la structure d'une fonction de modélisation (CODE NON EXECUTABLE)
2
3 my_model <- my_modeling_function(
4 formula = y ~ x + z,
5 data = my_data,
6 [other_parameters, ...]
7 )
Analysons ce code en détail. La ligne 3 consiste dans une simple association entre une référence symbolique, my_model, avec le résultat de l'exécution d'une fonction de modélisation, my_modeling_function(). Bien entendu, il existe une différence substantielle entre ces deux composantes :
- La référence symbolique
my_modelpeut avoir tout nom arbitraire que vous souhaitez utiliser, du moment qu'il respecte les règles syntaxique du langage - La fonction de modélisation, nommée ici
my_modeling_function(...), doit en revanche être une fonction existante, c'est-à-dire qu'elle est disponible dans l'environnement de la session de travail. Elle peut être à disposition de l'environnement de deux manières différentes :- Une fonction de modélisation déjà disponibles dans les paquets de R de base, comme par exemple
lm()out.test() - Une fonction chargée à travers un paquet externe qui la met à disposition dans l'environnement, comme par exemple les paquets car ou afex
- Une fonction de modélisation déjà disponibles dans les paquets de R de base, comme par exemple
La ligne 4 introduit le concept fondamental de formule. La formule s'occupe de formaliser, avec une syntaxe concise, la relation entre les données/variables du modèle. Dans le cas de notre exemple, la formule se lit de la manière suivante :
y ~ x + z
yreprésente la valeur que le modèle veut expliquer ou prédire. Selon l'approche utilisée à la modélisation, elle est souvent appelée variable dépendante (VD), variable expliquée, outcome ou explandandum- Le symbole
~(à lire tilde) peut se traduire avec la locution est expliqué/prédit par. Il sert donc à séparer la formule entre ce que nous souhaitons expliquer ou prédire, et les éléments utilisés pour effectuer l'explication ou la prédiction. Nous pouvons donc lire notre formule de la manière suivante y est expliqué/prédit par x et z. xetzsont par conséquent les valeurs qui expliquent ou prédisenty. Encore une fois, selon l'approche adoptée, on les identifie souvent en tant que variables indépendantes (VIs), prédicteurs ou explanans
y, x et z font références à des valeurs contenus dans la structure de données qui est passée en argument à la ligne 5, avec le code data = my_data. Par conséquent, my_data doit être une structure de données qui peut identifier des valeurs y, x et z (pas forcément dans cet ordre). Dans la plupart de cas, il s'agit des noms des colonnes d'une structure de données de type rectangulaire (e.g. un data.frame dans R de base ou un tibble dans l'écosystème tidyverse) :
| x | y | z | ... |
|---|---|---|---|
| x1 | y1 | z1 | ... |
| x2 | y2 | z2 | ... |
| ... | ... | ... | ... |
| xn | yn | zn | ... |
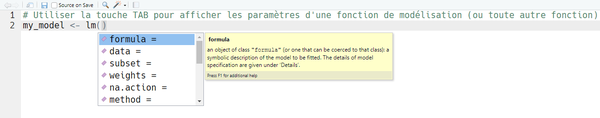
À la ligne 6, la notation conventionnelle [other_parameters, ...] signale que certains fonctions de modélisation acceptent des paramètres supplémentaires qui permettent de mieux adapter la modélisation aux besoins particulier de l'analyse. Ces paramètres dépendent évidemment de la fonction spécifique utilisées et il est souvent nécessaire de consulter la documentation ou des tutoriels pour connaître les différentes possibilités. Si vous utilisez RStudio, il est également possible de se servir de la touche TAB à l'intérieur des parenthèses de la fonction pour avoir un aperçu des paramètres acceptés, souvent avec une explication qui peut déjà aider un peu :
Différentes combinaisons dans la formule
La notation de la formule passée à la fonction doit évidemment s'adapter selon la modélisation à effectuer. On peut en effet combiner les éléments de différentes manières, en utilisant également d'autres symboles pour dénoter des rapports entre les éléments de la formule. Voici quelques exemples de notations qui sont valables dans plusieurs fonctions de modélisation, même dans des paquets externes. Pour brévité, nous utiliserons les termesde variable(s) dépendante(s) ~ variable(s) indépendante(s), abrégées en VD ~ VI, mais bien évidemment ces termes peuvent être substitués avec des combinaisons alternatives (e.g., outcome(s) ~ prédicteur(s), explenandum ~ explenans, ...). Pour l'instant nous nous limitons à afficher la syntaxe avec une référence au nom de tests classiques, souvent utilisés en sciences sociales, qui peuvent être rapportés à la formule. Des exemples concrets plus bas illustrerons les conséquences sur le modèle.
- y ~ x
- La VD est expliquée par une seule VI (e.g. t-test, One-way ANOVA, régression linéaire simple)
- y ~ x + z + c + d
- La VD est expliquée par les 4 VIs (e.g., régression linéaire multiple)
- y + a + b ~ x + z
- Les trois VDs y, a et b sont expliquées par les 2 VIs (e.g., MANOVA)
- y ~ x + z + x:z
- La VD est expliquée par les 2 VIs, plus un effet d'interaction/modération, dénoté par
x:z(e.g. ANOVA factorielle)
- La VD est expliquée par les 2 VIs, plus un effet d'interaction/modération, dénoté par
- y ~ x * z
- Même résultat du point précédent, mais en format raccourcis. L'utilisation de
*implique automatiquement l'effet d'interaction/modération.
- Même résultat du point précédent, mais en format raccourcis. L'utilisation de
- y ~ .
- Raccourcis pour dénoter que la VD est expliquée par toutes les VIs de la structure de données (i.e. toutes les autres colonnes)
- y ~ . - w
- Raccourcis pour dénoter que la VD est expliquée par toutes les VIs de la structure de données (i.e. toutes les autres colonnes) sauf la VI w.
Version courte de la fonction de modélisation
Comme il arrive souvent en R, les noms des paramètres utilisés très souvent dans une fonction peuvent ne pas être explicités, à condition d'en respecter l'ordre. Par conséquent, plusieurs fonctions de modélisation peuvent se passer des noms des paramètres formula = ... et data = .... Par exemple :
my_model <- my_modeling_function(y ~ x + z, my_data)
Le Output d'une fonction de modélisation
Contrairement aux logiciels d'analyse statistiques de type point-and-click (SPSS, Jamovi, ...) qui affichent souvent un grand nombre de résultats suite à une analyse (mais parfois pas ceux qui nous intéressent vraiment...), le Output d'une fonction de modélisation avec R est simplement un objet, souvent de type liste, avec lequel on peut interagir ultérieurement, surtout si nous l'avons stocké dans une référence symbolique.
En revanche, s'il existe plus ou moins une certaine cohérence dans les formats d'Input des fonctions de modélisation (i.e. formula + data + autres paramètres), le format de Output peut varier énormément d'une fonction à l'autre, ce qui est souvent source de frustration et incompréhension. Nous verrons dans la suite de cet article que cet aspect peut être géré par des paquets externes qui s'occupent de mettre en forme les résultats des fonctions de modélisations, mais pour l'instant focalisons nous sur un exemple de Output.
Dans le code suivant, nous simulons le score de 200 participants au jeu Super Mario Bros. Le score est généré aléatoirement selon une distribution normale avec la moyenne de 100 et l'écart type de 15. Chaque joueur a utilisé soit le personnage Mario, soit le personnage Luigi. Ensuite, nous utilisons deux fonctions de modélisation disponibles out of the box en R qui nous permettrons de comparer les différences dans l'Output :
- la fonction
lm(), qui correspond à une régression linéaire simple dans notre cas - la fonction
t.test()avec homogénéité de la variance, qui correspond à un test t de Student
Les deux modélisations sont attribuées respectivement aux références symboliques lm_model et t_model.
# Simulation de 200 scores M = 100, SD = 15, associés aléatoirement à deux personnages
game_scores <- data.frame(
score = rnorm(200, 100, 15),
player = sample(c("Mario", "Luigi"), 200, replace = TRUE)
)
# Régression linéaire simple
lm_model <- lm(
formula = score ~ player,
data = game_scores
)
# Student t-test (homogénéité de la variance)
t_model <- t.test(
formula = score ~ player,
data = game_scores,
var.equal = TRUE
)
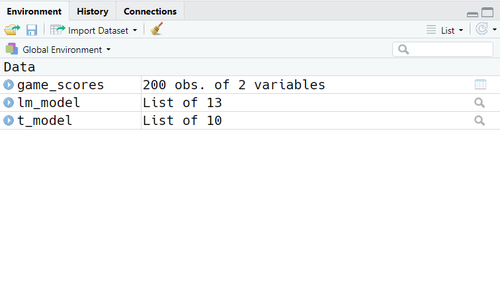
Lorsqu'on exécute ce bout de code, il n'y a aucun Output qui s'affiche à la console. En revanche, l'environnement global de votre session de travail se sera peuplé avec trois nouveaux éléments. Si vous utilisez RStudio, vous pouvez les noter dans le tab correspondant :
Nous pouvons noter que les deux Output des fonctions de modélisations sont deux listes, mais qu'elles diffèrent déjà dans le nombre d'éléments :
lm_modelest une liste avec 13 élémentst_modelest une liste avec 10 éléments
Il existe différents possibilités pour interagir avec ce type d'Output, par exemple :
- Accès direct à l'Output général
- Accès aux sous-éléments de l'Output
- Passer le Output en tant que Input pour une autre fonction
Accès direct à l'Output général
On peut accéder à l'Output général si on saisie tout simplement la référence symbolique à laquelle nous avons associé le résultat de la fonction de modélisation (ou alternativement si on invoque directement la fonction sans passer à travers une référence symbolique). Cet Output général est une sorte de résumé que les créateurs de la fonction décident de mettre à disposition comme représentatif du modèle, mais qui n'est pas le seule résultat accessible. Voyons cette explication plutôt abstraite appliquée aux deux fonctions de modélisations que nous avons utilisées. Si on saisie respectivement les deux références symboliques des modèles, nous obtenons à la console deux messages très différents :
lm_modelCall: lm(formula = score ~ player, data = game_scores) Coefficients: (Intercept) playerMario 99.77 2.84
t_modelTwo Sample t-test data: score by player t = -1.28, df = 198, p-value = 0.2 alternative hypothesis: true difference in means is not equal to 0 95 percent confidence interval: -7.2272 1.5508 sample estimates: mean in group Luigi mean in group Mario 99.766 102.604
Le Output de t_model est beaucoup plus informatif par rapport à lm_model, avec déjà plusieurs indicateurs corollaires (e.g. le test statistique t, les degrés de liberté, la p-valeur, ...).
Accès aux sous-éléments de l'Output
Les résultats d'une fonction de modélisation sont des objets de R comme tous les autres. Il est donc possible d'accéder aux différents sous-éléments qui peuvent souvent donner des informations spécifiques d'intérêt. De plus, ces éléments peuvent être ré-utilisé dans d'autres computations, ou référencés dans la création dynamique de report (voir Introduction à Rmarkdown).
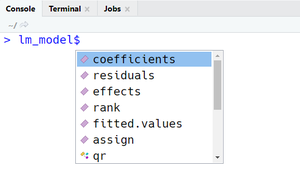
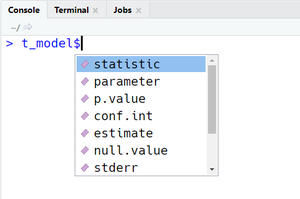
Dans nos exemples, il suffit d'ajouter le symbole $ de sélection d'un sous-élément dans une liste pour pouvoir accéder à des indicateurs des deux modèles. Si vous utilisez RStudio, la liste de sous-éléments va s'afficher à l'écran pour vous faciliter la tâche (voir Figures à côté). En alternative, on peut utiliser la fonction attributes() pour afficher à la console tous les sous-éléments et des informations complémentaires sur l'Ouput, comme par exemple sa classe :
attribues(lm_model)$names [1] "coefficients" "residuals" "effects" "rank" "fitted.values" [6] "assign" "qr" "df.residual" "contrasts" "xlevels" [11] "call" "terms" "model" $class [1] "lm"
attributes(t_model)$names [1] "statistic" "parameter" "p.value" "conf.int" "estimate" "null.value" [7] "stderr" "alternative" "method" "data.name" $class [1] "htest"
Les sous-éléments peuvent être à leur tours des structures de données, par exemple des listes ou des vecteurs, et peuvent ainsi être ultérieurement sélectionnés de manière plus précise. Par exemple, on peut accéder singulièrement aux intervalles de confiances autour du t.test() de la manière suivante :
- Limit inférieur :
t_model$conf.int[1] - Limit supérieur :
t_model$conf.int[2]
Passer le Output en tant que Input pour une autre fonction
L'Output d'une fonction de modélisation peut être passé en tant que Input à une autre fonction, qui s'occupe d'extraire différents indicateurs et les combiner. Par exemple, si on passe la référence symbolique à l'intérieur de la fonction summary() qui est souvent utilisée pour récupérer des informations depuis des structures de données ou listes, la situation par rapport à l'accès directe qu'on a vu plus haut est radicalement opposée :
summary(lm_model)Call: lm(formula = score ~ player, data = game_scores) Residuals: Min 1Q Median 3Q Max -43.98 -11.44 -0.79 11.24 42.86 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 99.77 1.64 61.00 <0.0000000000000002 playerMario 2.84 2.23 1.28 0.2 Residual standard error: 15.7 on 198 degrees of freedom Multiple R-squared: 0.00815, Adjusted R-squared: 0.00314 F-statistic: 1.63 on 1 and 198 DF, p-value: 0.204
summary(t_test)Length Class Mode statistic 1 -none- numeric parameter 1 -none- numeric p.value 1 -none- numeric conf.int 2 -none- numeric estimate 2 -none- numeric null.value 1 -none- numeric stderr 1 -none- numeric alternative 1 -none- character method 1 -none- character data.name 1 -none- character
Un autre exemple concerne la fonction plot() (voir Introduction à la visualization des données avec R), dont le comportement est encore plus différent selon le modèle que vous passez en argument :
plot(lm_model)- Cette utilisation de la fonction crée 4 graphiques qui sont souvent utilisés pour évaluer les postulats d'une regression linéaire, car elle est pré-programmée pour traiter un objet avec la classe
lm.
- Cette utilisation de la fonction crée 4 graphiques qui sont souvent utilisés pour évaluer les postulats d'une regression linéaire, car elle est pré-programmée pour traiter un objet avec la classe
plot(t_model)- Cette utilisation de la fonction déclenche une erreur car elle n'est pas pré-programmée pour traiter un objet avec la classe
htest.
- Cette utilisation de la fonction déclenche une erreur car elle n'est pas pré-programmée pour traiter un objet avec la classe
Le comportement en terme de Input/Output d'une fonction qui s'occupe à son tour des résultats d'une fonction de modélisation peut donc varier selon les intentions des créateurs de la fonction. Avec le temps ou selon les contextes, des nouveaux indicateurs peuvent être intéressants ou nécessaires. Pour cette raison, il existe des paquets externes qui peuvent être plus adaptés aux besoins.
Différentes approches à la modélisation des données
La modélisation des données est un sujet très vaste et complexe, qui implique plusieurs considérations philosophiques et méthodologiques qui dépassent le cadre introductif de cet article. Néanmoins, cette complexité se traduit inévitablement dans l'écosystème de R et il est donc utile de pouvoir l'aborder, au moins de manière superficielle.
Nous pouvons diviser les différences entre modélisations de données en deux grandes catégories :
- Différences philosophiques et méthodologiques relatives souvent à des interprétations du concept de probabilité différentes;
- Différences techniques au sein de la même approche philosophique/méthodologique dans l'implémentation pratique du même modèle.
Les deux catégories s'influencent mutuellement est sont destinées à changer dans le temps suite au débat et les indications de la communauté scientifique sur le sujet. En particulier dans ces dernières années, plusieurs propositions ont été faites pour modifier radicalement les pratiques dans l'analyse statistique dans les sciences sociales, notamment en psychologie. Nous proposons ici une liste non exhaustive de quelques propositions, en essayant de garder un regard neutre. L'utilité de cette liste est principalement celle de donner un aperçu de comment ces différences influencent profondément le fonctionnement, et par extension le choix, d'outils techniques de modélisation des données. Cette prémisse sert également à rendre attentif au fait que, parfois, la complexité de l'utilisation de R dans ce contexte relève principalement de la complexité du domaine lui-même. En d'autres termes, comparé à des logiciels de type point-and-click dans lequel les choix de l'utilisateur sont souvent guidées de manière plus ou moins stricte, avec R il existe une plus grande flexibilité, accompagnée inévitablement par une plus grand choix d'options et par conséquent d'incertitude potentielle.
Ne plus utiliser le terme significatif dans l'interprétation d'un test statistique
L'utilisation du term significatif serait devenu désormais trop ambigu, surtout à cause d'une mauvaise compréhension de la p-valeur qui accompagne l'interprétation de la significativité statistique. Une confusion entre la significativité statistique et la significativité pratique serait l'une des conséquences majeure à la base de cette proposition. Par exemple, il peut arriver de lire dans un article scientifique qu'une différence entre moyennes significative à p=0.00001 est retenue comme plus importante d'un point de vue pratique qu'une différence à p=0.02. Ou encore que la significativité d'un indicateur statistique dont la p-valeur est comprises entre 0.05 et 0.1 soit considérée tendancielle. Vous pouvez retrouver cette approche dans les résultats des tests statistiques classiques (voir plus bas) en R avec l'utilisation d'astérisques qui accompagnent les paramètres. La présence de ces astérisques, qui peut-être gérée à travers une option, est ironiquement identifiée par certains avec la pratique du stargazing (regarder les étoiles). Voici un exemple basé sur un jeu de donné déjà disponible en R de base avec la référence mtcars. L'exemple en soit est inintéressant si ce n'est pour la forme de l'output :
# Stargazing is ON
options(show.signif.stars = TRUE)
# Regréssion linéaire multiple :
# consommation carburant (mpg) ~ puissance (hp) + poids (wt) + boîte de vitesse automatique/manuelle (am)
lm_cars <- lm(formula = mpg ~ hp + wt + am, data = mtcars)
summary(lm_cars)
Le Output du sommaire est le suivant :
Call:
lm(formula = mpg ~ hp + wt + am, data = mtcars)
Residuals:
Min 1Q Median 3Q Max
-3.422 -1.792 -0.379 1.225 5.532
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 34.00288 2.64266 12.87 0.00000000000028 ***
hp -0.03748 0.00961 -3.90 0.00055 ***
wt -2.87858 0.90497 -3.18 0.00357 **
am 2.08371 1.37642 1.51 0.14127
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 2.54 on 28 degrees of freedom
Multiple R-squared: 0.84, Adjusted R-squared: 0.823
F-statistic: 49 on 3 and 28 DF, p-value: 0.0000000000291
La ligne de l'Ouput Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1,ainsi que les différents astérisques à côté des paramètres de la régression linéaire multiple, illustrent bien cette approche. La présence de ces astérisques est donc déterminée par le rôle philosophique ou méthodologique qui est donnée à la p-valeur, l'utilisation d'un seuil de significativité (le niveau alpha) fixe ou adapté selon l'étude, etc.