Déterminer la structure 3D d'une protéine biologiquement importante
Déterminer la structure 3D d'une protéine biologiquement importante
Procédure
Trouver quelques protéines pertinentes à vos cours
|
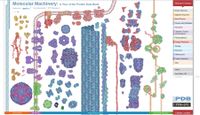
- choisir de préférence les structures 3D proposées à partir de ce poster MM PDB http://mm.rcsb.org/
- pour les protéines avec médicaments : choisir les structures 3D proposées par PDB (à partir de ce poster) http://pdb101.rcsb.org/browse/you-and-your-health
Nombreuses protéines avec leur structure et le fichier STL - prêtes pour impression
- Liste proposée au cours de formation continue PO 422 avec Dr. Marie-Claude Blatter du SIB
| Nom de la protéine ou du complexe | Lien vers UniProtKB/Swiss-Prot (section structure) | Lien vers l’entrée PDB (PDB AC)
Exemple de lien: https://www.rcsb.org/3d-view/5wrg |
Photo | Remarque | Fichiers .STL prêt à imprimer la plupart ajustées pour impression 3d par Julien Dacosta TECFA
ou Bertrand Emery (SEM Fablab) cf. exceptions dans le tableau |
|---|---|---|---|---|---|
| Hemoglobine Humaine | HBB_HUMAN HBA_HUMAN | 1a00
4hhb* 2hhb |
L’hémoglobine est constituée de 2 chaînes HBA et 2 chaînes HBB | Hemoglobin.stl | |
| COX1 + aspirine COX1 + ibuprofen | PGH1_SHEEP | 1pth*
1eqg |
Divers AINS (antidouleurs susceptibles de se fixer sur Cox1)
Fichiers fournis par Pr. Vincent Zoete, SIB Swiss Institute of Bioinformatics, University of Lausanne |
COX1-Protein | |
| Insuline Humaine | INS_HUMAN | 2hiu* 1ben | L’insuline est constituée de 2 chaînes (chaîne A : 21 aa, chaîne B : 30 aa). Dans l’entrée 2hiu, la chaîne B ne fait que 29 aa... | InsulineReady2print.stl | |
| Nucléosome (humain) | H4_HUMAN | 5b40 | La structure 3D contient les histones H4, H2A, H2B, H3.2 + ADN | 5B40_histone-protein-only.stl | |
| Nucléosome (batracien) | P62799 (H4_XENLA) | 1aoi* | H4, H2B11, H33C, H2A1 + ADN | ||
| Immuno-globuline IgG | GCAA_MOUSE IGH1M_MOUSE | 1igt* 1igy | IgG1-ready-2-print.stl.zip
Version avec cavités pour aimants permettant de simuler l'affinité des poches antigéniques (fichier imprimant 2 IgG1 tête-beche à la fois) | ||
| ATP Synthase | ATPB_BOVIN | 5ara* | Plusieurs sous-unités inclue ATP5B
Plus d’info : pdb101.rcsb.org/mot m/72 |
atp-synthase.stl.zip | |
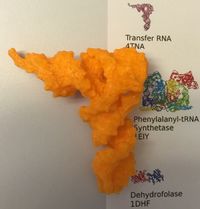
| tRNA | N'est pas une protéine ! | 4tna | yeast tRNAPhe | 4TNA-ready2print.stl | |
| TP53 + DNA | P53_HUMAN | 3q06 | Cf sur PDB | 393 aa : à l'heure de produire ce document aucune structure 3D ne couvre toute la séquence de la protéine | P53-avec-ADN.stl
(encore pas testé à l'impression) |
| RNA polymérase | RPAB4_YEAST | 2e2i* | Plusieurs sous-unités + DNA + RNA | ||
| Taq Polymérase | P19821 | 1TAU | 1TAU-Taq-polymerase.stl | ||
| Amylase | P12070 | 2die | 2die-amylase.stl | ||
| Protéine fluorescente de méduse | GFP_AEQVI | 1gfl | Séquence et structure complètes (blue GFP)
Green GFP |
1GFL_SingleChain-ribbon-thick.stl
| |
| Répresseur opéron lactose | LACI_ECOLI | 1lbh | Multimère de la même chaîne | lactose-operon-repressor-1lbh.stl | |
| CFTR
Protéine dont le défaut cause la mucoviscidose |
CFTR_HUMAN
P13569 |
5uak | cf. Scénario pour trouver la mutation la plus fréquente la ∂F508 cause la plus fréquente de la mucoviscidose) dans une puce à ADN (µ-array) | CFTR-ready-to-print.stl | |
| CRISPR-Cas9 | Q99ZW2 | 5F9R | La fermeture éclair de taille grossière se trouve en mercerie ou récupérer sur un habit / sac. | Cas9-ready-to-print.stl | |
| Spike protein du virus SARS-Cov-2 | P0DTC2 | 6VSB
6VXX |
image
|
Cf.processus de fabrication SEMlab | |
| Récepteur à l'acétycholine- | P02711 | 2bg9 | recepteur-nicotiniques.MP4 | 2bg9-acetycholine-recepteur.stl | |
| Erythropoétine EPO | P01588 | 1cn4 | 1cn4-EPO.stl |
Grand répertoired'exemples-proteines-3d-stl/
Ces structures ont été choisies en collaboration entre Dr. M.C.Blatter du SIB et F. Lombard (IUFE TECFA) et imprimées par Julien Dacosta, Vincent Widmer, Stephane Morand de TECFA, coordonnés par Prof. Daniel K. Schneider TECFA. Bertand Emery au Service d’impression 3D du SEMLab en a imprimé une partie. La COX1 et les AINS ont été fournis par Pr. Vincent Zoete, SIB Swiss Institute of Bioinformatics, University of Lausanne
Exemples de questions pour TP de biologie / pour s’en inspirer
- On dit parfois que la séquence d’acides aminés (a.a) détermine la fonction de la protéine. En quoi est-ce correct et en quoi cela est-il incomplet ?
- Comment la structure secondaire et tertiaire est-elle établie avec ce que vous avez pu voir jusqu’ici ? ( dans quels organites ? Comment ?)
- Peut-on actuellement prédire la forme que prendra une protéine à partir de sa séquence ?
- Comment détermine-t-on la forme que prend effectivement une protéine ?
- Comment la forme constatée détermine-t-elle l’activité de la protéine ?
- Comparez la séquence sur UniProtKB, puis la forme 3D pour diverses protéines:
- Insuline (http://www.rcsb.org/structure/1ben ; alternative (avec la séquence de la protéine: https://www.rcsb.org/3d-view/1ben/))
- Immunoglobuline IgG ( http://www.rcsb.org/structure/1igy; alternative (avec la séquence de la protéine: https://www.rcsb.org/3d-view/1igy/))
- Quelles parties de la forme de l’hormone, l’anticorps semble être en rapport avec leur fonction ?
- La forme détermine-t-elle seule la fonction ?
- Essayez de déterminer comment la forme d’un anticorps Ig détermine sa fonction ?
- Pour ces deux protéines, quelles parties de la protéine pourraient – à votre avis - changer un peu suite à une mutation sans gravement mettre en cause son fonctionnement et finalement réduire la fécondité de l'animal qui a ce génome-là ?
- Pour quelles autres parties un changement risque-t-il de nuire au fonctionnement de la protéine ?
- Idem pour l’histone HIST1H4A
- Idem pour l’insuline INS
- Idem pour le récepteur à la mélanotropine MC1R
- Concluez sur le lien entre forme et fonction, les limites du modèle « clé- serrure »
Liens externes
- Service d’impression 3D au SEMLab : dédié au DIP
Scénarios pédagogiques où il peut s'intégrer
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB
Les fichiers .STL ont été optimisées pour l'impression par Juline Dacosta (TECFA), sauf : la GFP par Romain Deweale.
La COX1 et les AINS ont été fournis par Pr. Vincent Zoete, SIB Swiss Institute of Bioinformatics, University of Lausanne