« Limite introns-exons pas discernable » : différence entre les versions
mAucun résumé des modifications |
|||
| (2 versions intermédiaires par 2 utilisateurs non affichées) | |||
| Ligne 1 : | Ligne 1 : | ||
== | |||
== prouver que la limite introns-exons n'est pas discernable aisément. == | |||
Un gène eucaryote est composé de plusieurs exons et introns. La structure d'un gène (nombre d'exons) est toujours la même, mais certains exons sont alternatifs, c'est-à-dire qu'ils ne sont pas présents dans tous les ARNm. L'épissage alternatif permet la production d'ARNm avec des combinaisons différentes d'exons. La définition d'un exon est donc parfois difficile à comprendre, puisque certains sont alternatifs: ils ne sont pas présents dans tous les ARNms, mais ce sont quand même des exons. C'est ce que ce scénario met en évidence avec l'aide des 'Genome browser'. 'Genome data viewer' permet de voir l'alignement des différents ARNm avec la séquence génomique. | |||
== Procédure == | == Procédure == | ||
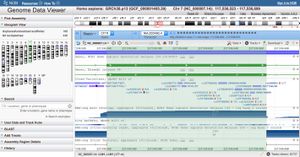
Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici][[Fichier:CFTR gene Exon7 highlith in GDV.jpg|alt=CFTR gene Exon7 highlith in GDV|vignette|CFTR gene Exon7 highlith in GDV|lien=https://edutechwiki.unige.ch/fr/Fichier:CFTR_gene_Exon7_highlith_in_GDV.jpg]] | Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici][[Fichier:CFTR gene Exon7 highlith in GDV.jpg|alt=CFTR gene Exon7 highlith in GDV|vignette|CFTR gene Exon7 highlith in GDV|lien=https://edutechwiki.unige.ch/fr/Fichier:CFTR_gene_Exon7_highlith_in_GDV.jpg]] | ||
*Sélectionner un gène (p. ex CFTR) selon | *Choisir l'espèce humaine | ||
*Sélectionner un gène (p. ex CFTR) selon scénario [[Séquence du gène de protéines sur chromosomes|trouver séquence des gènes d'une protéine]] ([https://www.ncbi.nlm.nih.gov/genome/gdv/browser/blast/?cfg=NCID_1_32903492_130.14.18.128_9146_1584116293_2168709450 solution]) | |||
*Observer les bandes bleues plus bas sous ''RNAseq exon coverage'' et ''RNAseq intron spanning read'' | *Observer les bandes bleues plus bas sous ''RNAseq exon coverage'' et ''RNAseq intron spanning read'' | ||
*Chercher à voir si la limite est bien distincte ou si il y a des zones qui sont parfois exprimées comme 'exons' ou non (des zones qui ne sont retrouvées que dans certains mRNAs). | *Chercher à voir si la limite est bien distincte ou si il y a des zones qui sont parfois exprimées comme 'exons' ou non (des zones qui ne sont retrouvées que dans certains mRNAs). | ||
*Discuter de l'épissage alternatif | *Eventuellement Discuter de l'épissage alternatif | ||
== ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | == ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | ||
Synthèse d'un élève : La limite introns-exons est difficilement discernable car sur le graphique, on n'observe qu’une partie des exons déborde sur une partie des introns, il est donc compliqué d’apercevoir une limite car celle-ci n’est pas clair et bien délimitée. On éprouve donc que les introns peuvent être des exons et les exons peuvent être des introns en fonction de la protéine produite. Avec l’épissage alternatif, la limite introns-exons est donc | Synthèse d'un élève : La limite introns-exons est difficilement discernable car sur le graphique, on n'observe qu’une partie des exons déborde sur une partie des introns, il est donc compliqué d’apercevoir une limite car celle-ci n’est pas clair et bien délimitée. On éprouve donc que les introns peuvent être des exons et les exons peuvent être des introns en fonction de la protéine produite. Avec l’épissage alternatif, la limite introns-exons est donc floue. | ||
== Scénarios pédagogiques où il peut s'intégrer == | == Scénarios pédagogiques où il peut s'intégrer == | ||
A faire encore | A faire encore | ||
---- | ---- | ||
* | |||
===== Références ===== | |||
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB | |||
Retour à [[Bioinformatique : opportunités pour l’enseignement]] | Retour à [[Bioinformatique : opportunités pour l’enseignement]] | ||
[[Catégorie: BioInfoScenarios]] | [[Catégorie: BioInfoScenarios]] | ||
Dernière version du 23 septembre 2022 à 10:31
prouver que la limite introns-exons n'est pas discernable aisément.
Un gène eucaryote est composé de plusieurs exons et introns. La structure d'un gène (nombre d'exons) est toujours la même, mais certains exons sont alternatifs, c'est-à-dire qu'ils ne sont pas présents dans tous les ARNm. L'épissage alternatif permet la production d'ARNm avec des combinaisons différentes d'exons. La définition d'un exon est donc parfois difficile à comprendre, puisque certains sont alternatifs: ils ne sont pas présents dans tous les ARNms, mais ce sont quand même des exons. C'est ce que ce scénario met en évidence avec l'aide des 'Genome browser'. 'Genome data viewer' permet de voir l'alignement des différents ARNm avec la séquence génomique.
Procédure
Ouvrir Genome data Viewer GDV ici
- Choisir l'espèce humaine
- Sélectionner un gène (p. ex CFTR) selon scénario trouver séquence des gènes d'une protéine (solution)
- Observer les bandes bleues plus bas sous RNAseq exon coverage et RNAseq intron spanning read
- Chercher à voir si la limite est bien distincte ou si il y a des zones qui sont parfois exprimées comme 'exons' ou non (des zones qui ne sont retrouvées que dans certains mRNAs).
- Eventuellement Discuter de l'épissage alternatif
Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe
Synthèse d'un élève : La limite introns-exons est difficilement discernable car sur le graphique, on n'observe qu’une partie des exons déborde sur une partie des introns, il est donc compliqué d’apercevoir une limite car celle-ci n’est pas clair et bien délimitée. On éprouve donc que les introns peuvent être des exons et les exons peuvent être des introns en fonction de la protéine produite. Avec l’épissage alternatif, la limite introns-exons est donc floue.
Scénarios pédagogiques où il peut s'intégrer
A faire encore
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB