« Génome pas constitué de gènes seulement » : différence entre les versions
(mise en palce de la séquence) |
m (→Procédure) |
||
| (3 versions intermédiaires par le même utilisateur non affichées) | |||
| Ligne 5 : | Ligne 5 : | ||
Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici] | Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici] | ||
Suivre ce [[Séquence du gène de protéines sur chromosomes|scénario]] | Suivre ce [[Séquence du gène de protéines sur chromosomes|scénario]] | ||
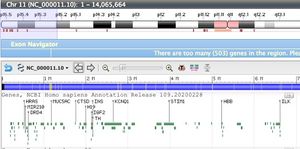
[[Fichier:Ins-zoomout-hbb-in-GDV.jpg|alt=zoom sur un fragment du chr 11 montrant les gènes ( autour de INS ) |vignette|zoom sur un fragment du chr 11 montrant les gènes ( autour de INS ) ]] | |||
Choisir l'espèce humaine, taper CFTR <small>( ou un autre exemple EPO, INS, AMY1A, HIST1H4A, CFTR, GH1, MC1R, TP53, ATP5F1A, AVPR1A, COL1A1, CYTB, OPN1LW, RHO , GULOP (pseudogène). )</small> | |||
Dézoomer (loupe - ) ou zoomer (loupe +) pour avoir une vue d’ensemble d' un large part du chromosome étudié - on voit la zone en grisé bleu s'élargir, aller jusqu'à au moins un dixième, mais en visualisant la totalité c'est encore plus visible. | |||
Observer les gènes (indiqués par des barres) et les zones qui ne sont pas des gènes. | |||
Estimer la proportion de l'ADN qui est des gènes | |||
Ces zones d'ATCG sont-elles codantes au sens vu en classe ? | |||
Trouver d'autres gènes connus | |||
P. ex si on est parti de INS (Chr11: 2,159,779 - 2,161,209 ) on peut trouver HBB (hemoglobin subunit beta) : dézoomer et à droite trouver la région 5,200 k zoomer sur la position (Chr11: 5,225,464 - 5,227,071) [https://www.ncbi.nlm.nih.gov/genome/gdv/browser/genome/?cfg=NCID_1_26088728_130.14.18.128_9146_1663924700_672495646 solution] <small>(en date du 23.09.22)</small> | |||
== ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | == ''Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe '' == | ||
| Ligne 23 : | Ligne 25 : | ||
== Scénarios pédagogiques où il peut s'intégrer == | == Scénarios pédagogiques où il peut s'intégrer == | ||
* | |||
===== Références ===== | |||
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB | |||
Retour à [[Bioinformatique : opportunités pour l’enseignement]] | Retour à [[Bioinformatique : opportunités pour l’enseignement]] | ||
[[Catégorie: BioInfoScenarios]] | [[Catégorie: BioInfoScenarios]] | ||
Dernière version du 23 septembre 2022 à 10:20
Éprouver qu'une grande partie du génome n'est pas constituée de gènes
Procédure
Ouvrir Genome data Viewer GDV ici
Suivre ce scénario
Choisir l'espèce humaine, taper CFTR ( ou un autre exemple EPO, INS, AMY1A, HIST1H4A, CFTR, GH1, MC1R, TP53, ATP5F1A, AVPR1A, COL1A1, CYTB, OPN1LW, RHO , GULOP (pseudogène). )
Dézoomer (loupe - ) ou zoomer (loupe +) pour avoir une vue d’ensemble d' un large part du chromosome étudié - on voit la zone en grisé bleu s'élargir, aller jusqu'à au moins un dixième, mais en visualisant la totalité c'est encore plus visible.
Observer les gènes (indiqués par des barres) et les zones qui ne sont pas des gènes.
Estimer la proportion de l'ADN qui est des gènes
Ces zones d'ATCG sont-elles codantes au sens vu en classe ?
Trouver d'autres gènes connus
P. ex si on est parti de INS (Chr11: 2,159,779 - 2,161,209 ) on peut trouver HBB (hemoglobin subunit beta) : dézoomer et à droite trouver la région 5,200 k zoomer sur la position (Chr11: 5,225,464 - 5,227,071) solution (en date du 23.09.22)
Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe
Lorsqu’on dézoome pour avoir une vue d’ensemble du chromosome où le gène se situe ( en général, pour tous les gènes étudiés par la classe, on remarque que les gènes ne constituent qu’une infime partie du génome). Entre les gènes, on retrouve des séquences non codantes. Il existe donc des bases ATCG qui ne codent pas.
Scénarios pédagogiques où il peut s'intégrer
Références
Scénario établi en partie sur la base des indications scientifiques de M.-C. Blatter du SIB