Introduction à Tidyverse
| Pensée computationnelle avec R | |
|---|---|
| ⚐ à finaliser | ☸ débutant |
| ⚒ 2023/01/11 | |
| Catégorie: R | |
Introduction
Tidyverse (Wickham et al., 2019) est une collection de paquets de R qui se caractérise par le choix délibéré d'appliquer certains principes, ou bonnes pratiques, au Data Science. Les paquets qui font partie de l'écosystème de Tidyverse implémentent ces principes à travers le partage d'une syntaxe similaire, uniformité dans les formats de Input et Output des fonctions, et une philosophie commune, inspirée par la notion de Tidy data (Wickham, 2014). La flexibilité et possibilité d'extension de l'écosystème Tidyverse font ainsi que les paquets puissent être utilisés indifféremment par des novices en R ou des utilisateurs expérimentés, pour des projets simples ou complexes.
Dans cette page nous proposons un aperçu de cet écosystème, surtout d'un point de vue conceptuel. Les paquets principaux qui composent le Tidyverse sont ensuite abordés de manière plus spécifiques dans une liste de tutoriels sur ce wiki ou à travers une liste des ressources externes.
Note sur la version
Cette page se réfère à la version 1.3.x de Tidyverse (voir versionnage sémantique). Même si l'écosystème des paquets liés au Tidyverse est constamment en évolution, les informations contenues dans cet article devrait être valable dans la plupart des cas également pour des versions successives.
Crédits
L'écosystème lié à Tidyvese est en général très ouvert et attentif aux novices/débutants en R. Il existe par conséquent des nombreuses ressources dans le web - surtout en anglais - disponibles sous Licence Creative Commons qui prévoit l'utilisation libre, pour des finalités non commerciales, avec attribution de la source. Cet article reprend plusieurs concepts et quelques exemples, en les adaptant au contexte d'intérêt de ce wiki, depuis :
- R for Data Science de Garrett Grolemund et Hadley Wickham. Il existe une traduction française du livre sous le titre R pour les data sciences.
Prérequis
Aucune connaissance préalable n'est nécessaire pour suivre le contenu de cet article. Cependant, pour maximiser sa compréhension et la possibilité d'appliquer directement les éléments traités, les articles suivants peuvent être très utils :
L'utilisation de RStudio est aussi recommandée, même si le code peut être exécuté dans tout environnement qui supporte le langage R. Pour plus d'information, voir mise en place d'un environnement de travail avec R.
Installation et incorporation
Tidyverse est une collection de différents paquets de R qui peuvent être installés et incorporé dans vos scripts de deux manières différentes :
- Installation/incorporation globale à travers le paquet
tidyverse - Installation/incorporation ponctuelle des paquets singulièrement (voir plus bas pour la liste)
La phase d'installation est d'incorporation peuvent être distinctes, c'est-à-dire que vous pouvez installer le paquet global et ensuite incorporer singulièrement un ou plusieurs paquets. Lorsque vous vous approchez pour la première fois à l'univers Tidyverse, il est conseillé d'utiliser le paquet global. Par la suite, par contre, il est plus indiqué d'incorporer seulement les paquets que vous utilisez, car le paquet global est assez lourd.
Installation et incorporation globale
Pour installer tous les paquets de Tidyverse utilisez la commande suivante :
install.packages("tidyverse")
Pour charger et incorporer Tidyverse dans vos scripts, utilisez :
library(tidyverse)
Lorsque vous lancez cette commande, vous aller voir dans la console un message qui vous informe des paquets que vous avez chargés, ainsi que leur version actuelle. Par exemple :
-- Attaching packages --------------------------------------- tidyverse 1.3.0 --
v ggplot2 3.2.1 v purrr 0.3.3
v tibble 2.1.3 v dplyr 0.8.3
v tidyr 1.0.0 v stringr 1.4.0
v readr 1.3.1 v forcats 0.4.0
-- Conflicts ------------------------------------------ tidyverse_conflicts() --
x dplyr::filter() masks stats::filter()
x dplyr::lag() masks stats::lag()
Le message vous informe également d'éventuels conflits avec d'autres fonctions. Cet aspect dépasse le cadre introductif de cet article et il est commun à tout autre paquet de R. Néanmoins, il est bien de savoir que Tidyverse vous informe des conflits.
Incorporation individuelle d'autres paquets installés globalement
La commande install.packages("tidyverse") installe également d'autres paquets de l'univers Tidyverse qui ne sont en revanche pas incorporés automatiquement avec la commande library(tidyverse) afin de ne pas charger des éléments non utilisés par la suite. Pour incorporer ces paquets il faudra donc charger le paquet séparément, en plus du paquet global, e.g. :
# Charger le paquet global
library(tidyverse)
# Charger le paquet rvest pour faire du web scraping
library(rvest)
Le paquet rvest, qui sert pour faire du Web scraping (voir Web scraping avec R pour plus d'infos), est déjà installé à travers le paquet global tidyverse, mais contrairement à d'autres paquets comme ggplot2 ou dplyr, il n'est pas automatiquement chargé. Pour cette raison, il faut indiquer expressément son incorporation avec la commande library(rvest).
Installation et incorporation ponctuelle
Tout paquet de Tidyverse peut être également installé et/ou incorporé individuellement. Par exemple, pour installer le paquet ggplot2, qui permet de créer des représentations graphiques, la commande est la suivante :
install.packages("ggplot2")
Pour charger et incorporer le paquet dans vos script, la commande est la suivante :
library(ggplot2)
La philosophie Tidy
L'univers lié à Tidyverse part du constat que, en Data Science, la plupart du temps est consacrée au traitement, transformation, nettoyage et visualization des données. Il existe donc un déséquilibre entre la quantité de ressources disponibles sur la modélisation des données (e.g. statistiques inférentielles, modèles prédictifs, ...) et les ressources destinées à la préparation et manipulation des données en amont.
La partie qui précède la modélisation des données est souvent négligée dans les articles ou tutoriels qui la considère comme acquise. De même, les logiciels statistiques traditionnels ont souvent des fonctionnalités plutôt limitées d'un point de vue de la manipulation des données, car il s'attende à un format rectangulaire des données (divisées en ligne et colonnes) déjà propre et prêt pour l'analyse.
Par conséquent, les utilisateurs sont obligés d'utiliser des flux de traitement de données qui nécessitent de plusieurs logiciels, avec souvent des passages entre plusieurs formats de fichiers différents. De plus, ils doivent répéter cette procédure plusieurs fois si les données en leur possession changent entre temps, ce qui expose à des erreurs humains qui pourraient être évités avec un traitement plus automatisé des informations. Par exemple, il est une pratique assez fréquente de créer plusieurs copies des fichiers de données en fonction de certaines transformations qu'on peut appliquer aux données (e.g. filtrer certaines observations, créer des indices composées, etc.). Comme résultat, nous nous retrouvons rapidement avec plusieurs fichiers du style :
- data.xlsx
- data2.xlsx
- data3.xlsx
- data-transformed.xlsx
- ...
Ce fichiers sont fréquemment le résultat du copier/coller d'autres fichiers encore, par exemple récupérés depuis différentes sources (e.g. exportation d'un logiciel de questionnaires en ligne comme Qualtrics ou Limesurvey). Avec le temps et les différentes manipulations apportées, il peut s'avérer difficile, voire impossible, de remonter aux données originales qui ont entre temps été écrasées par des sauvegardes plus récentes.
Tidyverse met à disposition un écosystème de paquets qui ont l'objectif de faciliter et promouvoir des bonnes pratiques dans les différents stades du traitement des données en Data Science, avec une attention particulière aux phases initiales, afin que les données soient prêtes pour des processus successifs relatifs à la modélisation et à la communication. Pour ce faire, Tidyverse s'appuie sur des principes fondamentaux tels que :
- Uniformité dans les formats d'entrée et de sortie des données
- Faciliter le processus de traitement à travers des étapes séquentielles
- Adopter une approche fonctionnelle à la programmation
Uniformité des formats d'entrée et sortie des données
Le traitement des données en Data Science présuppose une transformation entre un état précédent la manipulation et un état successif. La transformation peut contribuer à modifier le format des données. Un exemple peut faciliter la compréhension de cet aspect qui est principalement technique, mais qui peut avoir des repercussions dans le codage :
# Les données d'entrée sont dans un data.frame
data <- data.frame(
participant = c("P1", "P2", "P3"),
score = c(25, 17, 29)
)
class(data) # donne "data.frame"
# Les données sont réduites dans à un seul indicateur, la moyenne de la colonne score du data.frame
avg_score <- mean(data$score)
class(avg_score) # donne "numeric"
Or, ce passage comporte le fait que la moyenne (i.e. avg_score) est maintenant dans un format différent des données d'entrée. Ce qui est compréhensible, car il s'agit d'un indicateur simple comparé à une structure de donnée. Néanmoins, il s'agit d'une décision arbitraire sur comment représenter les données, car même un indicateur simple peut être représenté dans une structure des données :
# Les données d'entrée sont dans un data.frame
data <- data.frame(
participant = c("P1", "P2", "P3"),
score = c(25, 17, 29)
)
class(data) # donne "data.frame"
# Les données sont maintenues dans un data.frame avec un seul indicateur
score <- data.frame(
avg = mean(data$score)
)
class(score) # donne "data.frame"
Avec cette approche différente, on maintient le "data.frame" comme structure de base de nos données, même si pour le score nous n'avons qu'un seul élément, auquel on a maintenant accès avec score$avg. Ce passage, qui est apparemment rébarbative, permet par contre d'avoir une uniformité dans les formats des données. De cette manière, nous sommes certaines qu'on peut les traiter avec toutes les manipulations qui peuvent s'appliquer aux données de type "data.frame".
L'unité de Tidyverse : le tibble()
Le format des données étant un aspect fondamentale, Tidyverse introduit un nouveau format, appelé tibble, qui vise reprendre les points forts des data.frame de R, en les améliorants ultérieurement. Pour apprécier conceptuellement les tibble il faudrait d'abord une certaine expérience avec les différents formats des données de R, surtout le data.frame. Mais ceci n'empêche pas de pouvoir en tout cas exploiter les bénéfices pratiques des tibble, dont nous proposons ici un bref survol.
Pour créer un nouveau tibble, on peut utiliser la fonction tibble() de la manière suivante :
my_tibble <- tibble(
participant = c("P1", "P2", "P3",
time = c(178, 165, 144),
)
Si on contrôle quel type d'élément est my_tible à travers la fonction class(), nous obtenons le message suivant :
class(my_tibble)
# [1] "tbl_df" "tbl" "data.frame"
Comme vous pouvez le voir, my_tibble garde la classe "data.frame", dont le tibble est effectivement une extension. Pour comparer les deux, nous allons également créer la même structure de donnés, mais avec la fonction de base data.frame() :
my_df<- data.frame(
participant = c("P1", "P2", "P3"),
time = c(178, 165, 144)
)
Vous pouvez déjà vous apercevoir des différences simplement avec la fonction print() :
print(my_tibble)
print(my_df)
La première donne :
# A tibble: 3 x 2
participant time
<chr> <dbl>
1 P1 178
2 P2 165
3 P3 144
La deuxième donne :
participant time
1 P1 178
2 P2 165
3 P3 144
On peut noter comme le format du tibble est plus ordonné et également plus informatifs, car il affiche le type de données contenues dans chaque colonne.
Il existe une fonction, as_tibble(), qui permet de faire le upgrade entre un data.frame et un tibble :
upgraded_df <- as_tibble(my_df)
Processus de traitement séquentiel
Un autre aspects central à la philosophie Tidy est le traitement séquentiel de l'information, ce qui se traduit concrètement avec l'utilisation du pipe %>%. Cet élément peut être déroutant au début, mais intégré à l'intérieur de l'écosystème Tidyverse il peut s'avérer très utile. Voici un simple exemple pour illustrer le principe :
execution_time <- c(120, 97, 111, 134, 123, 150)
# Calculer la moyenne "normalement"
mean(execution_time)
# Calculer la moyenne avec le pipe
execution_time %>% mean()
Ce bout de code propose deux manières différentes pour exécuter la même opération : calculer la moyenne d'une série (vecteur) de temps d'exécution d'un tâche quelconque. La première manière, traditionnelle, consiste à passer le vecteur en tant que argument à la fonction mean() :
mean(execution_time)
La deuxième manière, au contraire, modifie l'ordre en mettant d'abord l'objet execution_time, et ensuite la procedure mean() elle-même :
execution_time %>% mean()
Ce mécanisme est rendu possible à travers l'opérateur %>%, appelé pipe, et qui peut être interprété tout simplement comme l'équivalent en langage courant de et puis... :
- Prend la liste des temps d'exécution et puis... calcule la moyenne
À bien noter que la fonction mean() dans la variante du pipe n'a pas d'argument. Ceci s'explique par le fait que dans le traitement séquentiel rendu possible par le pipe, le résultat de l'étape précédente est passé automatiquement en argument de la fonction successive. Imaginons une situation qui nécessite de trois procédures séquentielles sur le même object, comme par exemple cuire des carottes :
- Éplucher les carottes :
eplucher(carottes) - Couper les carottes épluchées en rondelles :
couper(carottes_épluchées) - Cuire les carottes épluchées et coupées dans une casserole :
cuire(carottes_épluchées_coupées)
Dans du code traditionnelle on devrait écrire le code pour obtenir le résultat final de cette manière :
cuire(couper(eplucher(carottes)))
C'est-à-dire exactement à l'envers par rapport à l'exécution temporelle des procédures. Avec l'opérateur pipe, au contraire, le code est le suivant :
carottes %>% eplucher() %>% couper() %>% cuire()
Dans cette variante, la fonction successive reçoit en argument le résultat de l'étape précédente, donc :
- eplucher() reçoit en argument les carottes nature
- couper() reçoit en argument le résultat de eplucher(carottes)
- cuire() reçoit en argument le résultat de couper(eplucher(carottes))
On peut complexifier cet exemple en considérant que les fonctions couper() et cuire() peuvent avoir d'autres arguments, notamment :
couper(quoi, comment)cuire(quoi, comment, combien_de_minutes)
À ce moment, notre chaîne d'exécution du pipe peut ressembler à ceci :
carottes %>% eplucher() %>% couper("à rondelles") %>% cuire("à la vapeur", 10)
Pour faciliter la lecture du code, vous verrez que souvent le code Tidyverse est divisé en plusieurs lignes, avec le pipe comme dernier élément de chaque ligne :
carottes %>%
eplucher() %>%
couper("à rondelles") %>%
cuire("à la vapeur", 10)
Nous pouvons ainsi comparer les deux variantes :
# Code traditionnel
carottes_traditionnelles <- cuire(couper(eplucher(carottes)), "à rondelles"), "à la vapeur", 10)
# Tidyverse
carottes_a_la_tidyverse <- carottes %>%
eplucher() %>%
couper("à rondelles") %>%
cuire("à la vapeur", 10)
Pour faciliter l'adoption du pipe, il existe un raccourci en RStudio qui permet de l'insérer dans le code source :
- Pour Windows :
Ctrl + Shift + M - Pour Mac :
Cmd + Shift + M
|>. Les deux opérateurs %>% et |> peuvent être utilisés dans la plupart des cas de manière interchangeable. Programmation fonctionnelle
Le concept de programmation fonctionnelle dépasse le contexte d'un article introductif, car il se réfère à un paradigme de programmation, dont la compréhension nécessiterait d'ailleurs de la comparaison avec d'autres paradigmes (e.g. programmation par objets). Mais on peut néanmoins introduire ce concept important dans l'écosystème de Tidyverse à travers le principe suivant : en Tidyverse les données sont manipulées avec des fonctions qui se traduisent par des verbs. Parmi les verbs les plus utilisés dans Tidyverse, transformés en fonction, on retrouve :
select()pour isoler une ou plusieurs colonnes d'une structure de donnéesfilter()pour isoler une ou plusieurs lignes d'une structure de donnéesarrange()pour ordonner les lignes d'une structure de données selon un critère spécifiquegroup_by()pour grouper les lignes d'une structure de données en fonction d'un critère spécifiquesummarise()pour agréger les lignes d'une structure de données selon une computation spécifique (e.g. moyenne, ...)
Même sans avoir encore vu du code qui utilise ces fonctions, on peut déjà noter qu'elles sont caractérisée par une nomenclature sémantique qui est proche de leur utilité réelle. De plus, toutes ces fonctions acceptent en tant que premier paramètre la structure de données à manier, ce qui permet de le combiner avec le traitement séquentiel décrit dans le point précédent. On retrouve par conséquent très souvent en Tidyverse des bouts de code similaires à celui-ci :
promoted_students <- students %>%
select(first_name, last_name, grade) %>%
filter(grade >= 4) %>%
arrange(last_name, first_name)
La lecture de cette manipulation peut se faire assez facilement :
- Ligne 1 : on manipule la structure des données
studentspour créer une référence symbolique avec la liste des étudiants qui ont réussit le cours, nomméepromoted_students - Ligne 2 : parmi toutes les colonnes qui composent la structure, nous sommes intéressés aux colonnes
first_name,last_name, etgrade - Ligne 3 : nous appliquons un filtre qui retient seulement les lignes dont la valeur à l'intérieur de la colonne
gradeest égale ou supérieur à 4, ce qui est correspond au barème en Suisse - Ligne 4 : enfin, on ordonne les étudiants par leur nom de famille, et ensuite prénom en cas d'homonymie
La philosophie du tidy data
Toute la philosophie de Tidyverse est construite autour de l'organisation des données. En effet, cet aspect est souvent sous-estimé ou géré par des mythes dont l'origine est parfois incertaine ou lié à l'utilisation spécifique de certains logiciels d'analyse statistique. Dans cette section, nous proposons un bref exemple conceptuel qui compare deux approches différentes :
- Le mythe du tableau omni-compréhensive
- L'approche Tidy
Nous verrons les mêmes données dans les deux perspectives afin de mettre en évidence les différences. Par la suite de l'article, nous verrons comment passer d'un format à l'autre avec une extension de cet exemple de base, ainsi que les avantages de passer à l'approche Tidy.
Le mythe du tableau omni-compréhensive
Un mythe assez fréquent en sciences sociales consiste dans le tableau rectangulaire qui englobe toutes les données des participants, représenté dans l'exemple suivant :
| participant | age | faculty | task1 | task2 | task3 | question1 | question2 | question3 |
|---|---|---|---|---|---|---|---|---|
| P1 | 27 | Computer Science | 162 | 120 | 200 | 1 | 2 | 1 |
| P2 | 38 | Psychology | 97 | 126 | 185 | 5 | 4 | 3 |
| P3 | 22 | Other | 150 | 177 | 170 | 7 | 6 | 7 |
Ce tableau présente en réalité différentes informations. On retrouve :
- Des informations personnelles comme l'âge ou la faculté d’appartenance
- Des informations relatives à des mesures sur trois tâches, par exemple le temps en seconds nécessaire pour mener à bien la tâche correspondante
- Des informations relatives à un questionnaire d'évaluation sur une échelle ordinale de 1 à 7
Nous avons limité à trois tâches et à trois questions pour des raisons de brévité, mais ce type de tableaux peuvent rapidement s'étaler en largeur à plusieurs dizaines de colonnes. À l'occurrence, d'ailleurs, le nombre de colonnes peut être ultérieurement augmenté avec des indices composés, comme par exemple les moyennes des tâches et des questions :
| participant | age | faculty | task1 | task2 | task3 | question1 | question2 | question3 | avg_tasks | avg_questions |
|---|---|---|---|---|---|---|---|---|---|---|
| P1 | 27 | Computer Science | 162 | 120 | 200 | 1 | 2 | 1 | 160.6667 | 1.333333 |
| P2 | 38 | Psychology | 97 | 126 | 185 | 5 | 4 | 3 | 136.0000 | 4.000000 |
| P3 | 22 | Other | 150 | 177 | 170 | 7 | 6 | 7 | 165.6667 | 6.666667 |
Les mêmes données en format Tidy
Les mêmes données peuvent être organisées de manière différentes, en suivant notamment les 3 principes de base du format Tidy (Wickham, 2014, p. 4) :
- Chaque variable forme une colonne
- Chaque observation forme une ligne
- Chaque unité d'observation forme un tableau
Pour mieux approfondir ces principes conceptuels plus tard, nous proposons d'abord des exemples concrets basés sur les mêmes données du tableau omni-compréhensive de la section précédente. Nous avons déterminé que ce tableau contient 3 unité d'observations différentes :
- Des données personnelles
- Des données relatives à la performance à des tâches
- Des données relatives à l'évaluation de la tâche à travers un questionnaire
Voici l'organisation Tidy de ces trois tableaux.
Tableau avec données personnelles
Le tableau avec les données personnelles est assez intuitif même au format Tidy, car il présuppose une organisation similaire au tableau omni-compréhensive avec les observations/lignes représentées par les participants, mais avec les colonnes/variables limitées aux variables personnelles age et faculty :
| participant | age | faculty |
|---|---|---|
| P1 | 27 | Computer Science |
| P2 | 38 | Psychology |
| P3 | 22 | Other |
Tableau avec données relatives aux tâches
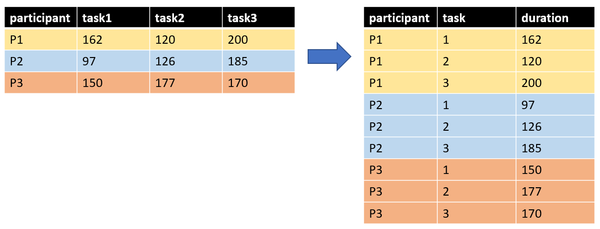
On pourrait appliquer le même principe pour les données relatives aux tâches, c'est-à-dire extraire simplement les colonnes relatives aux trois tâches de la manière suivante :
| participant | task1 | task2 | task3 |
|---|---|---|---|
| P1 | 162 | 120 | 200 |
| P2 | 97 | 126 | 185 |
| P3 | 150 | 177 | 170 |
Cependant, nous pouvons considérer que les trois tâches sont en réalité trois observations différentes, même si elle partage un élément commun, c'est-à-dire le participant. En plus, cette organisation ne permet pas vraiment de comprendre à quoi se réfère le chiffre dans chaque tâche. Par conséquent, un format plus informatif consiste à organiser les données de cette manière :
| participant | task | duration |
|---|---|---|
| P1 | 1 | 162 |
| P1 | 2 | 120 |
| P1 | 3 | 200 |
| P2 | 1 | 97 |
| P2 | 2 | 126 |
| P2 | 3 | 185 |
| P3 | 1 | 150 |
| P3 | 2 | 177 |
| P3 | 3 | 170 |
Cette variante du tableau comporte seulement 3 colonnes, mais en revanche il se compose de 9 lignes, qui sont le résultat de la multiplication de 3 participants par les 3 tâches. On parle dans ce cas du passage d'un format large à un format long. L'image ci-bas représente le même mécanisme à l'aide de couleurs qui permettent de mieux retracer le passage des données.
Le format long est plus difficile à lire pour l'oeil humain. En revanche, ce type de tableau est beaucoup plus simple à traiter par un ordinateur, qui peut plus facilement filtrer, agréger, ou calculer des indices composés, comme on le verra plus bas dans la page.
Tableau avec évaluation du questionnaire
Le même principe peut s'appliquer aux données issues du questionnaire, dont les données peuvent passer du format large
| participant | question1 | question2 | question3 |
|---|---|---|---|
| P1 | 1 | 2 | 1 |
| P2 | 5 | 4 | 3 |
| P3 | 7 | 6 | 7 |
au format long :
| participant | item | evaluation |
|---|---|---|
| P1 | 1 | 1 |
| P1 | 2 | 2 |
| P1 | 3 | 1 |
| P2 | 1 | 5 |
| P2 | 2 | 4 |
| P2 | 3 | 3 |
| P3 | 1 | 7 |
| P3 | 2 | 6 |
| P3 | 3 | 7 |
Agrégation des observations
L'un des avantages du format long - on en verra d'autres dans la section suivant - consiste dans le fait que le même tableau peut être agrégé de manières différentes en fonction de la colonne qu'on utilise pour grouper les observations. Par exemple, le tableau des tâches peut :
- Agréger les observations par participant
- Agréger les observations par le numéro de la tâche
| participant | task | duration |
|---|---|---|
| P1 | 1 | 162 |
| P1 | 2 | 120 |
| P1 | 3 | 200 |
| P2 | 1 | 97 |
| P2 | 2 | 126 |
| P2 | 3 | 185 |
| P3 | 1 | 150 |
| P3 | 2 | 177 |
| P3 | 3 | 170 |
Dans l'agrégation par participant, les lignes sont regroupées en fonction de la première colonne. On peut par exemple déterminer la moyenne (mean_duration) et l'écart type (sd_duration) pour chaque participant :
| participant | number_of_tasks | mean_duration | sd_duration |
|---|---|---|---|
| P1 | 3 | 160.6667 | 40.01666 |
| P2 | 3 | 136.0000 | 44.84417 |
| P3 | 3 | 165.6667 | 14.01190 |
Dans l'agrégation par tâche, les lignes sont regroupées par la deuxième colonne :
| task | number_of_participants | mean_duration | sd_duration |
|---|---|---|---|
| 1 | 3 | 136.3333 | 34.58805 |
| 2 | 3 | 141.0000 | 31.32092 |
| 3 | 3 | 185.0000 | 15.00000 |
Vous pouvez noter comme les deux tableaux maintiennent exactement la même structure en termes de colonnes. De plus, ces colonnes resteraient les mêmes indépendamment du nombre de participants ou du nombre des tâches ; seulement le nombre des liges en serait affecté.
En même temps, le nombre des colonnes pourrait être incrémenté en fonction de nouveaux indices, comme par exemple la valeur minimale ou maximale, l'écart, etc.
Tutoriels Tidyverse
Dans ce wiki
Pour approfondir les concepts traités dans cet article de manière appliquée et découvrir les différents possibilités mises à disposition par l'écosystème de Tidyverse, ce wiki propose les tutoriels suivants :
- : modalités d'importation de données alternatives à l'importation de base de R, ainsi que des fonctions pour exporter les données
- : comment préparer un ou plusieurs jeux de données pour faciliter la manipulation, visualisation et modélisation
- : comment effectuer des opérations récurrentes dans le traitement des données (filtrer, agréger, créer des nouvelles variables, ...) sur une ou plusieurs tableaux
- : créer des graphiques et autres visualisations des données adaptées aux objectifs exploratoires ou communicationnels.
- Web scraping avec R (rvest): utiliser le paquet rvest pour automatiser l'extraction de données depuis le web.
Ressources/tutoriels externes
Tidyverse est un écosystème très utilisé dans différents domaines d'application de R. Il existe donc un nombre de ressources et tutoriels basés sur les paquets de Tidyverse. Voici une sélection de ressources et tutoriels, surtout en anglais :
- Introduction à R et au tidyverse : introduction exhaustive avec des nombreuses illustrations écrite par Julien Barnier
- R for data science : livre en accès libre sur le web, écrit par Hadley Wickham et Garrett Grolemund, qui propose un aperçu générale de tidyverse. Le livre est destiné principalement à un public novice en R
- Statistical Inference via Data Science. A ModernDive into R and the Tidyverse : livre en accès lobre sur le web, écrit par Chester Ismay et Albert Y. Kim, qui utilise Tidyverse plus spécifiquement dans le domaine de la modélisation des données d'un point de vue inférentiel
Références
- Wickham, H., & Grolemund, G. (2016). R for data science: Import, tidy, transform, visualize, and model data. O’Reilly Media, Inc.
- Wickham, H. (2014). Tidy Data. Journal of Statistical Software, 59(10). https://doi.org/10.18637/jss.v059.i10
- Wickham, H., Averick, M., Bryan, J., Chang, W., McGowan, L., François, R., … Yutani, H. (2019). Welcome to the Tidyverse. Journal of Open Source Software, 4(43), 1686. https://doi.org/10.21105/joss.01686