Analyses statistiques avec R
Donato Cereghetti, 2013
Introduction
R est un langage de programmation et un environnement open-source permettant le traitement des données et les analyses statistiques. Sur cette page nous proposons un tutoriel permettant de s’initier à cet environnement. Dans un premier temps, nous présenterons les bases du fonctionnement de R. Par la suite, nous introduirons le traitement des données alphanumériques (analyse qualitative). Dans un troisième temps nous introduirons le traitement des données numériques (analyse quantitative). Enfin, nous nous focaliserons sur la génération et l'analyse des graphes et réseaux.
Dans cette page nous proposons un tutoriel pratique permettant d'introduire le lecteur au logiciel R.
Voir aussi:
Pourquoi préférer R à d'autres logiciels de traitement de données ?
- Il s’agit d’un logiciel gratuit à code source ouvert
- Il est compatible avec les systèmes Windows, MAC OS et Linux
- Il s’agit d’un logiciel très puissant et complet : en gros, il permet de tout faire !
- Il est en essor permanent
- En cas de problème, des milliers d'utilisateurs sont prêts à vous aider ! Cliquez ici pour accéder à une communauté française assez active
- La syntaxe est relativement simple, et s'apparente beaucoup à celle de Matlab
- R est très stable
Limites de R
- L'interface standard n'est pas user-friendly. L’application R Studio et l’interface graphique R-Commander permettent de surmonter cette limite.
Publique cible
Le public cible de R est constitué par toute personne désirant manipuler, traiter et analyser des données numériques ou alphanumériques.
D’après cereghd0, il ne faut pas avoir des compétences informatiques particulières pour apprendre à utiliser R. Cela dit, des connaissances de base en programmation faciliteraient la tâche.
Remarquons enfin que ce logiciel est très répandu dans le milieu académique.
Présentation du logiciel R
Installation de R
Installer sur Windows et Mac Pour installer R dirigez-vous sur http://cran.r-project.org/ et télécharger l’environnement convenant au système d’exploitation de votre ordinateur. Ensuite, installer aussi un "front-end" (voir ci-dessous).
Installer sur Ubuntu Installer R sur Linux est un petit peu plus compliqué, dans Debian/Ubuntu ce package n'est pas dans la liste de distribution par exemple.
Tips pour Ubuntu:
- Il faut vérifier l'archive de téléchargement, par exemple éditer à la main le fichier
/etc/apt/sources.list. Les Suisses mettront:
deb http://stat.ethz.ch/CRAN/bin/linux/ubuntu trusty/
- Notez le "trusty" s'applique aux installations Ubuntu 14LTS ! Suivez les instructions, sur http://cran.r-project.org/
- Radicalement tuer peut-etre une ancienne version:
sudo apt-get --purge remove r-base
- Installer
sudo apt-get update sudo apt-get install r-base
Installer un front-end plus convivial
On conseille également d'installer RStudio, une interface à R améliorée. TStudio améliore par exemple la gestion des scripts (vos séquences d'instruction) et des packages (extensions).
Introduction à l'environnement R
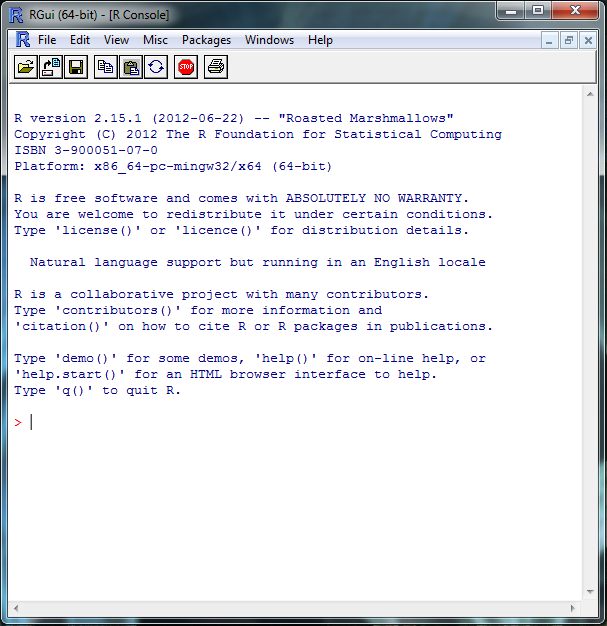
Voici comment l’environnement R se présente une fois l’application ouverte :
L’interface de l’environnement R se présente par une fenêtre contenant une barre de menus, une barre d’outils et la console R. Cette dernière permet d’introduire des commandes R (ou expressions).
L’interprétateur de R traduit et exécute les commandes introduites par l’utilisateur.
Le prompt de R est représenté par le symbole > . Ce caractère signifie que l’interprétateur de R attend l’introduction d’une commande de la part de l’utilisateur.
Remarquons que l’interface standard graphique de R paraît élémentaire et peu ergonomique. Plusieurs interfaces graphiques existent et peuvent être installées. Nous recommandons l’installation de R-Commander et R-Studio.
Voici une copie d'écran de R-Studio:
Installation de paquets
La plupart des paquets (extensions) de R sont disponibles à travers R. Exemple "statnet", un logiciels d'analyse de réseaux.
Install.packages("statnet")
- ou plus verbeux:
Install.packages("statnet", dependencies = TRUE)
Alternativement, si vous utilisez RStudio, cliquez sur l'onglet "Packages", puis "Install". Dans la fenêtre popup on peu ensuite indique le nom (ou le début du nom) du package...
L'aide et la documentation de R
La fonction help.start() permet d’acceder à l’aide en ligne de R.
help.start()
La fonction help() ou l'abréviation ? permet d'obtenir de l’aide sur des commandes R spécifiques. Exemples :
help(help) # lance la documentation de la fonction « help » ?mean # lance la documentation de la fonction « mean »
La fonction help.search() ou l'abréviation?? permet de chercher la documentation
??tm # lance une recherche pour "tm"
La fonction help.library() liste les paquet.
Tapez help(help) pour l'aide complète sur help
Commentaires
Le symbole # permet d'ajouter des commentaires dans R. Tout ce qui est écrit après ce symbole n'est pas interprété par R. Prenons l’exemple suivant :
3 + 2 # Ceci est un commentaire... remarquez que R interprète ce qui vient avant le symbole # et nous donne le résultat de l'addition
Les commentaires sont très utiles en programmation pour décrire ce que le code fait.
Les objets de R
Les objets sont des entités qui sont stockées dans la mémoire de R. Les opérateurs d’assignation <- et -> permettent de créer et de stocker des objets dans la session de R courante. Exemples :
Poids <- 80 # L’objet Poids reçoit la valeur 80 80 -> Poids # L’objet Poids reçoit la valeur 80
Il est important de créer des objets afin de pouvoir les manipuler dans un deuxième moment. Par exemple :
a <- 32+10
b <- 10*21
c <- a + b
Ainsi, si l’utilisateur ne crée pas des objets ; R exécute la commande et affiche l'output à l’écran. Cela dit, ce dernier ne sera pas accessible pour des traitements ultérieurs. Exemple :
32+10
10*21
# Contrairement à l’exemple ci-dessus, ici il est impossible de calculer la somme entre (32+10) et (10*21).
Remarquons que si on ne crée pas des objets, R traite les données... mais ces résultats seront inaccessibles pour des traitements succéssifs.
Types et structures de données
Dans R, différents types d'objets peuvent être crées et traités. Voici les principaux objets :
a <- NULL # a est un objet nul, vide
b <- TRUE # b est un objet logique, booléen
c <- 101 # c et Poids sont des objets numériques
Poids <- 85
d <- "Bonjour à tout le monde!" # d est un objet alphanumérique (il contient une chaîne des caractères)
Pour connaître le mode (type) d'un objet, utilisez la commande mode(). Ci dessous montrée avec les résultats affichés par R.
> mode(Poids)
[1] "numeric"
> mode(a)
[1] "NULL"
> mode(b)
[1] "logical"
> mode(c)
[1] "numeric"
> mode(d)
[1] "character"
Pour afficher la valeur d'un objet, on peut utiliser la fonction print(). Altérnativement on peut introduire tout simplement le nom de l’objet à afficher :
print(Poids) Poids print(a) a
Pour lister les objets existant dans la session de R, utilisez la fonction ls() ou objects() :
ls() objects()
Enfin, on peut supprimer des objets avec la fonction rm() :
rm(Poids) # L'objet Poids est supprimé rm(a) # L’objet a est supprimé rm(b, c, d) # Les objets b, c et d sont supprimés
Dans les prochaines sections, nous présenterons les objets fondamentaux de R : les vecteurs, les matrices et les data-frames.
Les vecteurs
Un vecteur est un objet atomique représentant une "ligne" de données: tous ses éléments sont caractérisés par le même mode (ou type). Plusieurs méthodes peuvent être utilisées pour générer un vecteur numérique. Le plus important est le collecteur c() :
c(3, 4, 1, 3) # vecteur numérique de longueur 4
Altérnativement, vous pouvez utiliser l'opérateur : , la fonction seq et la fonction rep :
1:15
seq(1,5, by = 0.2)
seq(1,5, length=10)
rep(1,10)
rep(1:3, times = 3)
rep(1:3, each = 3)
rep(1:3, times = 3, each = 2)
Pour générer un vecteur alphanumérique, utilisez le collecteur c() associé à des guillemets :
c('H', 'F', 'F', 'H', 'F')
Alternativement, vous pouvez utiliser la fonction rep() :
rep('H', 20)
Pour générer un vecteur logique, utilisez les opérateurs >, >=, <, <=, ==, != :
10 == (2+1) # Donne FALSE car 10 est différent de 3 10 != 3 # Donne TRUE car 10 est différent de 3 10 >= 7 # Donne TRUE car 10 est supérieur égal à 7 1:10 == 6 # Donne un vecteur logique de longueur 10. La valeur TRUE apparaît quand la condition est satisfaite ; la valeur FALSE apparaît quand la condition n'est pas satisfaite
Rappelons que les opérateurs <- et -> permettent de sauvegarder des objets dans la mémoire de R. Créons un vecteur numérique Age et un vecteur alphanumérique Sexe à travers les commandes suivantes :
Age <- c(19, 31, 28, 18, 25, 23, 27, 20)
Sexe <- c('F', 'H', 'F', 'H', 'H', 'F', 'H', 'F')
Affichons enfin les deux vecteurs avec les commandes :
Age Sexe
Pour sélectionner une partie d'un vecteur, utilisez l'opérateur de sélection [ ]. Etudiez les exemples suivants :
X = 10:20 X[3] # Donne le troisième élément du vecteur X X[c(6,9)] # Donne le sixième et neuvième élément du vecteur X X[3:5] # Donne le troisième quatrième et cinquième élément du vecteur X X[7:5] # Donne le septième, le sixième et le cinquième élément du vecteur X X[-c(4,7)] # Donne X sans le quatrième et septième élément du vecteur X X[-(1:3)] # Donne X sans les trois premiers éléments X[(X<15)] # Donne les éléments de X inférieur à 15 X[(X<17) & (X>13)] # Donne les éléments de X inférieurs à 17 ET supérieurs à 13 X[(X<13) | (X>17)] # Donne les éléments de X inférieurs à 13 OU supérieurs à 17
Les matrices
La matrice est un objet atomique et représente des tables à deux ou plusiers dimensions: tous ses éléments sont caractérisés par le même mode (ou type). Pour créer une matrice utilisez la fonction matrix() :
a1 <- matrix(1:6, ncol=2) # a1 est une matrice 3x2 a2 <- matrix(1:6, nrow=2) # a2 est une matrice 2x3 b1 <- matrix(1:6, ncol=2, byrow = TRUE) # b1 est une matrice 3x2 b2 <- matrix(1:6, nrow=2, byrow = TRUE) # b2 est une matrice 2x3
Pour sélectionner une partie d'une matrice, utilisez l'opérateur de sélection [ ] :
a1[3,2] # Donne l'élément de la matrice a1 qui se situe à la coordonnée [3,2] c'est-à-dire à la troisième ligne et deuxième colonne a1[2,] # Sélectionne la deuxième ligne de la matrice a1 a1[,2] # Sélectionne la deuxième colonne de la matrice a1 a1[-1,] # Matrice a1 sans sa première ligne a1[,-1] # Matrice a1 sans sa première colonne a1[-1,2] # Sélectionne la deuxième colonne de la matrice a1 en excluant la première ligne a2[,a2[1,]>2] # Sélectionne les colonnes de la matrice a2 pour lesquelles la valeur sur la première ligne est supérieure à 2
Les data frames
Le data-frame est un tableau des données à 2 dimensions dont les colonnes sont des variables de différents modes. Il s'agit de l'objet qu'il faut privilégier pour la recherche en sciences humaines. En effet, dans la plupart des cas, les bases des données sont composées par des variables qualitatives et quantitatives ! La fonction data.frame() permet de créer cet objet :
Age <- c(19, 31, 28, 18, 25, 23, 27, 20)
Sexe <- c('F', 'H', 'F', 'H', 'H', 'F', 'H', 'F')
Database <- data.frame(Age, Sexe) # Database est un data-frame comportant une variable qualitative et une variable quantitative
Database
Pour sélectionner une variable particulière d’un data-frame, vous pouvez utiliser le symbole $ :
Database$Age Database$Sexe
De manière analogue aux matrices, vous pouvez sélectionner une partie d’un data-frame en utilisant les opérateurs de sélection [ ] :
Database[1,2] Database[1:4,2]
Les listes
Les listes représentent les structures de données les plus souples. On peut créer des listes dans des listes qui ne doivent pas avoir la même longeur.
my_list_simple = list ( c(1,2,3), c("a","b"))
donne:
> my_list_simple 1 [1] 1 2 3 2 [1] "a" "b"
my_list = list ( liste_une = c(1,2,3), liste_deux= c("a","b"))
donne:
> my_list $liste_une [1] 1 2 3 $liste_deux [1] "a" "b"
Charger des jeux des données de R
La fonction data() permet de récupérer des bases des données qui existent dans R afin de les manipuler, analyser, traiter dans un deuxième moment :
data() # Permet d'afficher les bases des données de R pouvant être récupérées data(iris) # Permet de charger en mémoire la base des données « iris ». help(iris) # Lance l’aide associée à la base des données « iris ». ls() # Remarquons l’existence de l’objet « iris », qui a été chargé en mémoire iris
La fonction de ces bases de données est strictement pédagogique : elles permettent à l’utilisateur de tester aisément les différentes fonctions et commandes de R sur différents bases des données. Par exemple, nous pouvons résumer l'information contenue dans l'objet iris avec la commande suivante :
summary(iris)
Dans les prochains chapitres, nous chargerons des jeux des données de R dans le but d'introduire les différentes fonctions de R.
Importer des bases des données
Les fonctions read.table() et read.csv() permettent de lire et importer des fichiers .txt et .csv.
R peut directement lire un fichier depuis un URL.
L'intégration de la fonction file.choose() permet à l'utilisateur de sélectionner un fichier.
# Le fichier data.txt est lu est stocké dans un nouveau objet R nommé Database
Database <- read.table("data.txt", header = TRUE)
# Le séparateur utilisé dans le fichier délimité est la virgule
Database <- read.table(file.choose(), header = TRUE, sep = ",")
# Fichier de type CSV. Le séparateur utilisé dans le fichier csv est le point-virgule
Database <- read.csv(file.choose(), header = TRUE, sep= ";")
# Fichier de type CSV depuis un serveur web (ce fichier contient des stats de google webmaster tools pour edutechwiki ...)
Database <- read.csv("http://tecfa.unige.ch/guides/R/data/edutechwiki-fr-gw-oct-6-2014.csv", header = TRUE, sep= ",")
# Fichier de type Excel qui contient une simple matrice, la première ligne contient les noms de variables
library(xlsx)
Database <- read.xlsx("c:/dks/myexcel.xlsx", 1)
Visualiser les tableaux:
- Dans RStudio, cliquer sur le variable (par ex. "Database" ci-dessus) dans le panneau Environment
- Sinon, utilisez: summary(DB), dim(Database), etc.
Voir aussi:
- Importing Data (Quick-R)
Traitement des données alphanumériques
Dans un premiers temps nous explorerons les fonctions de R permettant de manipuler les objets de nature numérique de manière élémentaire. Dans la deuxième partie de ce chapitre, nous présenterons des méthodes quantitatives pouvant être appliquée à l’analyse de discours. Si ce dernier sujet vous intéresse, lisez aussi Text mining avec R. Ci-dessous on ne s'intéresse qu'aux fonctionnalités standardes de R, mais pas aux extensions.
Importation de texte
En règle générale, on conseille d'utiliser les fonctions des paquets "text mining", car elle permettent (a) de lire un ensemble de fichiers dans une structure de données "corpus" et (b) d'appliquer certains filtres dès le départ. Toutefois pour des analyses simples on peut utiliser les fonctionalités de base.
scanest une fonction flexible pour lire des données dans un vecteur.
Example:
#Lire un fichier caractère par caractère, et séparer le contenu ligne par ligne.
text.vector_char <- scan("http://edutechwiki.unige.ch/fmediawiki/api.php?action=parse&page=Civilization&format=xml",
what="character", sep="\n")
#Vérification
text.vector_char
readLineslit un fichier ligne par ligne
#Lire un fichier ligne par ligne
text.vector_lines <- readLines("http://edutechwiki.unige.ch/fmediawiki/api.php?action=parse&page=Civilization&format=xml")
#Vérification
text.vector_lines
- Les 2 exemples donnent (en principe) le même résultat, car on a utilisé scan avec un séparateur de lignes:
scan ("...", sep="\n")
read.tableetread.csvpermettent de lire des text en format tabulaire- voir ci-dessous...
Manipulations de bas niveau
Concaténation
Pour concaténer, juxtaposer des chaînes de caractères utiliser la fonction paste() :
paste("Bonjour", "!!!")
paste ("Bonjour", "!!!", sep = "_")
paste ("Bonjour", "!!!", sep = "")
x <- c("a", "b", "c")
paste(x,x)
paste(x,x, sep = "_")
paste(x,x, collapse="++")
paste(x,x, sep = "_", collapse="++")
Longueur des chaînes de caractères
Pour accéder à la longueur de chaînes utiliser la fonction nchar()
Texte1 <- "Bonjour!"
nchar(Texte1, type = "chars")
Texte2 <- c("Bonjour", "à", "vous", "tous", "!")
nchar(Texte2, type = "chars")
Extraire/remplacer un segment de chaîne
La fonction substr() permet d’extraire un segment de chaîne à partir des attributs « start » et « stop » :
Texte3 <- c("ABCDEFGHI")
substr(Texte3, start = 3, stop = 6)
Texte4 <- c("ABCD", "EFGH", "ILMN", "OPQR")
substr(Texte4, start = 2, stop = 3)
Les opérateurs d’assignation <- et -> permettent de remplacer des segments de chaîne :
substr(Texte3, start = 3, stop = 6) <- "@@@" Texte3 substr(Texte4, start = 2, stop = 3) <- "##" Texte4
La fonction substring() permet d’extraire un segment de chaîne à partir des attributs « first » et « last » :
Texte5 <- paste(LETTERS, collapse="")
substring(Texte5, first = 2)
Texte6 <- c("ABCD", "EFGHILMNOPQR")
substring(Texte6, first = 2)
Les opérateurs d’assignation <- et -> permettent de remplacer des segments de chaîne :
substring(Texte5, first = 3, last = 6) <- "@@@" Texte5
Transformations sur une chaîne
La fonction tolower() permet de transformer une chaîne de caractère en minuscule :
tolower("BoNjoUr!")
tolower(LETTERS)
La fonction toupper() permet de transformer une chaîne de caractère en majuscule :
toupper("BoNjoUr!")
toupper(letters)
La fonction chartr() permet de remplacer des lettres dans une chaîne des caractères :
Texte7 <- "cHaInE BiZarRe" chartr(Texte7, old = "caE", new = "¦@#") # La lettre « c » est remplacée par le symbole ¦, la lettre « a » est remplacée par le symbole @ et la lettre « E » est remplacée par le symbole #.
Scinder une chaîne des caractères
La fonction strsplit() permet de diviser une chaîne de caractères :
Texte8 <- c("J'aime jouer au football", "Je n'aime pas jouer à hockey sur glace")
strsplit(Texte8, split = " " , fixed = TRUE)
strsplit(Texte8, split = "'" , fixed = TRUE)
strsplit(Texte8, split = "a" , fixed = TRUE)
strsplit(Texte8, split = c("o", "u"), fixed = TRUE)
L’output de la fonction strsplit() est représentée par une liste. Vous pouvez utiliser la fonction unlist() pour créer un vecteur contenant tous les éléments de la liste :
unlist(strsplit(Texte8, split = " " , fixed = TRUE))
Rechercher des pattern
La fonction grep() permet de rechercher les éléments d’une liste qui présentent un pattern donné
Texte9 <- c("argue", "sténo", "huons", "remet", "ponce", "mites", "ligie", "vitre", "fluée", "floué", "ahana", "fonte", "boche", "tinté", "frime", "tente", "cédez", "votif", "ligné", "acéré")
grep(pattern = "a" , Texte9, value = FALSE, fixed = TRUE) # Donne les indices des éléments de Texte9 qui contiennent la lettre « a »
grep(pattern = "a" , Texte9, value = TRUE, fixed = TRUE) # Donne les éléments de Texte9 qui contiennent la lettre « a »
Les expressions régulières
Il existe plusieurs symboles qui permettent de faciliter les recherches dans les chaînes de caractères. Il s’agit notamment de :
- ^ : début d’une chaîne
- $ : fin d’une chaîne
- . : tout caractère
- | : disjonction (ou)
- [] : ensemble de caractères à égaler
- [^]: ensemble de caractères à ne pas égaler
- \\ : permet de rechercher des caractères spéciaux (« ? », « $ », « ^ »,…)
- * : 0 fois ou plus l’expression concernée
- + : 1 fois ou plus l’expression concernée
- ? : 0 ou 1 fois l’expression concernée
- {n} : nombre d’apparition de l’expression concernée
- {n,}: l’expression concernée apparaît n fois ou plus
- {,n}: l’expression concernée apparaît n fois ou moins
- () : regroupement
Pour rechercher dans une chaîne de caractères des expressions régulières avec la fonction grep, il faut fixer l’attribut « fixed » à la valeur FALSE :
Texte9 <- c("argue", "sténo", "huons", "remet", "ponce", "mites", "ligie", "vitre", "fluée", "floué", "ahana", "fonte", "boche", "tinté", "frime", "tente", "cédez", "votif", "ligné", "acéré")
grep(pattern = "^a", Texte9, value = TRUE, fixed=FALSE) # Donne les mots qui commencent par la lettre « a »
grep(pattern = "e$", Texte9, value = TRUE, fixed=FALSE) # Donne les mots qui se terminent par la lettre « e »
grep(pattern = "te", Texte9, value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence « te »
grep(pattern = "n.e", Texte9 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence « n.e »
grep(pattern = "na|ce" , Texte9 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence « na » ou la séquence « ce »
grep(pattern = "n.e|c.e" , Texte9 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence « n.e » ou la séquence « c.e »
grep(pattern = "[cdt]e" , Texte9 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence « ce » ou « de » ou « te »
grep(pattern = "[^cdt]e" , Texte9 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la lettre « e » et qui ne présentent pas les séquences « ce », « de » et « te »
Texte10 <- c("ab","abc", "ababc", "ababab", "abababc" ,"bab","cbabab","abba","abbc","ca","abaabc", "ababbc", "acac")
grep(pattern = "ab*c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : a(b)*c
grep(pattern = "ab+c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : a(b)+ c
grep(pattern = "ab?c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : a(b)? c
grep(pattern = "ab{2}c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : abbc
grep(pattern = "(ab)*c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : (ab)*c
grep(pattern = "(ab)+c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : (ab)+ c
grep(pattern = "(ab)?c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : (ab)? c
grep(pattern = "(ab){2}c" , Texte10 , value = TRUE, fixed=FALSE) # Donne les mots qui contiennent la séquence : ababc
Remplacer des pattern
La fonction gsub() permet de rechercher et remplacer un pattern dans un objet alphanumérique :
Texte9 <- c("argue", "sténo", "huons", "remet", "ponce", "mites", "ligie", "vitre", "fluée", "floué", "ahana", "fonte", "boche", "tinté", "frime", "tente", "cédez", "votif", "ligné", "acéré")
gsub(pattern = "o" , replacement = "O" , Texte9, fixed = TRUE)
gsub(pattern = "e" , replacement = "E" , Texte9, fixed = TRUE)
Traitement des données numériques
Les opérateurs arithmétiques classiques
La somme : +
3+5
La soustraction : -
5-1
La multiplication : *
3*2
La division : /
8/2
L’exposant : ^
3^3
La racine carrée : sqrt()
sqrt(9)
Le modulo (donne le reste d’une division) : %%
10%%3
Représenter des données sur R
L’objectif de ce chapitre est d’apprendre à utiliser les fonctions : hist(), density(), lines(), boxplot(), barplot(), pie(),mosaicplot(), assocplot(), plot(), abline(), pairs(), scatterplot3d().
Histogramme et fonction de densité
L’histogramme permet de représenter la distribution d’une variable continue. Vous pouvez construire des histogrammes avec la fonction hist() :
data(iris) hist(iris$Sepal.Length, prob = FALSE, main = "Histogramme de la longueur des sépales", xlab = "Longueur des sépales", ylab = "Effectif ", col = "lightblue") # Histogramme en effectif hist(iris$Sepal.Length, prob = TRUE, main = "Histogramme de la longueur des sépales", xlab = "Longueur des sépales", ylab = "Densité de la distribution", col = "lightblue") # Histogramme en densité
La fonction density() permet d’obtenir un estimateur à noyau de la densité :
plot(density(iris$Sepal.Length))
Pour tracer l'histogramme et la densité en même temps on peut utiliser le code suivant :
hist(iris$Sepal.Length, prob = TRUE, main = "Histogramme de la longueur des sépales", xlab = "Longueur des sépales", ylab = "Densité de la distribution", col = "lightblue") # Histogramme en densité lines(density(iris$Sepal.Length),col="red")
Boite à moustache
La boîte à moustache est une méthode rapide pour représenter et résumer le profil d’une variable continue. Pour créer une boîte à moustache utilisez la fonction boxplot() :
A <- rnorm(1000, 100, 10) # A est un vecteur de 1000 valeurs tirées d'une distribution gaussienne de moyenne 100 et d'écart-type 10
B <- rnorm(1000, 120, 20) # B est un vecteur de 1000 valeurs tirées d'une distribution gaussienne de moyenne 120 et d'écart-type 20
boxplot(A, main = "Distribution des valeurs générés")
boxplot(list(A, B), main = "Distribution des valeurs générés" , names = c("µ = 100, sd = 10", "µ = 120, sd = 20 "))
Diagramme en tuyaux d’orgue
Le diagramme en tuyaux d’orgue permet de représenter graphiquement la distribution d'une variable discrète. Pour créer un diagramme en tuyaux d’orgue, utilisez la fonction barplot() :
ventes <- c(200, 102, 32, 120, 310, 152)
names(ventes) <- c("Pomme","Cerise", "Abricot", "Poire", "Framboise", "Melon")
barplot(ventes, main = "Diagramme des ventes", col = "lightblue")
barplot(ventes, main = "Diagramme des ventes", col = "lightblue", horiz = TRUE)
Diagramme circulaire
Le diagramme circulaire permet de représenter des variables discrètes. Utilisez la fonction pie() pour construire des diagrammes circulaires :
ventes <- c(200, 102, 32, 120, 310, 152)
names(ventes) <- c("Pomme","Cerise", "Abricot", "Poire", "Framboise", "Melon")
pie(ventes, main = "Diagramme des ventes", col = rainbow(length(ventes)))
Diagramme empilé
Le diagramme empilé permet de représenter graphiquement les effectifs d’un tableau de contingence. Le diagramme empilé s’obtient au moyen de la fonction barplot() en fournissant un objet de mode matrix :
data(HairEyeColor) HairEye <- apply(HairEyeColor, c(1, 2), sum) barplot(HairEye, legend = rownames(HairEye), main = "Diagramme empilé", xlab = "Couleur des yeux")
Diagrammes en mosaïque
Le diagramme en mosaïque permet de représenter graphiquement les effectifs d’un tableau de contingence. De ce fait, la surface des mosaïques est proportionnelle aux effectifs observés dans les cellules du tableau de contingence. Pour créer un diagramme en mosaïque utilisez la fonction mosaicplot() :
mosaicplot(HairEye, main = "Diagramme en mosaïque", xlab = "Cheveux", ylab = "Yeux")
Graphique d’association
Le graphique d’association de Cohen-Friendly indique les déviations par rapport à l’indépendance dans une table de contingence. Utilisez la fonction assocplot() pour créer un graphique d’association :
assocplot(HairEye, main = "Graphique d’association", xlab = "Cheveux", ylab = "Yeux")
Nuage des points 2D
La fonction plot() permet – entre d’autres – de représenter deux variables continues dans un plan cartésien. Exemple :
data(USArrests) plot(USArrests$Murder~USArrests$Assault, main = " Nuage de points", xlab = "Nombre d’agressions", ylab = "Nombre d’assassinats") abline(lm(USArrests$Murder~USArrests$Assault), col = "red")
Remarque : la fonction “lm” permet de créer des modèles linéaires (on y reviendra).
Matrice des scatter plots
La fonction pairs() permet de représenter un scatter plot pour chaque couple possible de colonnes d’un dataframe ou d’une matrice donnée :
data <- USArrests[,c(1,2,4)] pairs(data)
Nuage des points 3D
La fonction scatterplot3d() permet de représenter trois variables dans un espace tridimensionnel. Pour utiliser cette fonction il faut installer le package « scatterplot3d » :
library(scatterplot3d) s3d <- scatterplot3d(USArrests$Assault, USArrests$Rape, USArrests$Murder, type= "h", highlight.3d = TRUE, angle = 45, scale.y = 0.5, pch = 20, main = "Nuage des points 3D", xlab = "Nombre d’agressions", ylab = " Nombre de viols", zlab = "Nombre d’assassinats") s3d$plane3d(lm(USArrests$Murder ~ USArrests$Assault + USArrests$Rape))
Corrgram
La fonction corrgram() permet de représenter graphiquement une matrice de corrélation de manière efficace. Pour utiliser cette fonction il faut installer le package « corrgram » :
library(corrgram) data(auto) corrgram(auto, order=TRUE, main="Auto data (PC order)")
Traitements statistiques élémentaires
L’objectif de ce chapitre est d’apprendre à utiliser les fonctions : mean(), sd(), median(), quantile(), min(), max(), summary() et cov().
Mesures de tendance centrale, de dispersion et de position
Les fonctions mean(), sd() restituent la moyenne, et respectivement l’écart-type d’un objet donné :
mean(USArrests$Murder) sd(USArrests$Murder)
Les fonctions median() et quantile() restituent le médian et les quantiles désirées :
median(USArrests$Murder) quantile(USArrests$Murder, c(0.25, 0.5, 0.75)) # Donne les 3 quartiles quantile(USArrests$Murder, c(0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9)) # Donne les 9 déciles
Les fonctions min() et max() restituent la valeur minimale et maximale d’un objet :
min(USArrests$Murder) max(USArrests$Murder)
Résumé statistique
La fonction summary() fournit une série d’information qui varie en fonction de l’objet d’input :
summary(iris) summary(USArrests)
Matrice de variance-covariance
La fonction var() restitue la matrice de variance-covariance d’une matrice ou d’un data-frame :
var(USArrests)
Approches corrélationnels
Corrélation
La fonction cor() restitue la matrice de corrélation d’une matrice ou d’un data-frame :
cor(USArrests)
Test sur un coefficient de corrélation
La fonction t.test() permet de tester la significativité d’un coefficient de corrélation donnée :
x <- c(44.4, 45.9, 41.9, 53.3, 44.7, 44.1, 50.7, 45.2, 60.1) y <- c( 2.6, 3.1, 2.5, 5.0, 3.6, 4.0, 5.2, 2.8, 3.8) cor.test(x,y)
Régression linéaire simple
La fonction lm() permet de bâtir de modèles de régression linéaire simple :
plot(USArrests$Murder~USArrests$Assault, main = " Nuage de points", xlab = "Nombre d’agressions", ylab = "Nombre d’assassinats") LinMod4 <- lm(USArrests$Murder~USArrests$Assault) summary(LinMod4) # Paramètres du modèle anova(LinMod4) abline(LinMod4, col= "red")
Un modèle linéaire nécessite la normalité des résidus et l'homoscédasticité. Utilisez le code suivant pour vérifier si ces deux conditions sont remplies :
par(mfrow = c(2,2)) plot(LinMod4, col.smooth = "black") # Analyse des résidus
Régression linéaire multiple
La fonction lm() permet de bâtir de modèles de régression linéaire multiple :
data <- USArrests[,c(1,2,4)] pairs(data) LinMod5 <- lm(USArrests$Murder ~ USArrests$Assault + USArrests$Rape) summary(LinMod5) # Paramètres du modèle anova(LinMod5)
Un modèle linéaire nécessite la normalité des résidus et l'homoscédasticité. Utilisez le code suivant pour vérifier si ces deux conditions sont remplies :
par(mfrow = c(2,2)) plot(LinMod5, col.smooth = "black") # Analyse des résidus
Statistiques inférentielles : t-test, Analyse de variance
Test de T sur un échantillon unique
La fonction t.test() permet – entre d’autres – de comparer une moyenne observée à une moyenne théorique.
Test bilatéral:
notes <- c(4, 4, 4.5, 3, 5, 6, 5.5, 5, 4, 3.5, 3, 5.5, 4, 6, 5, 5.5) m.theo <- 4.5 t.test(notes, mu = m.theo)
Test unilatéral droit:
m.theo <- 4 t.test(notes, mu = m.theo, alternative = "greater")
Test unilatéral gauche:
m.theo <- 5 t.test(notes, mu = m.theo, alternative = "less")
Ce test nécessite la normalité des données de l'échantillon. Vous pouvez la tester avec le code suivant :
shapiro.test(notes)
Test de T sur deux échantillons indépendants avec variance égales
La fonction t.test() permet – entre d’autres – de comparer deux moyennes issues de deux échantillons indépendants avec variances égales.
Test bilatéral:
A <- c(3,4,5,4,3,4,4,4,5,3,3,6,1,2) B <- c(5,7,6,4,5,3,5,5,3,6,5,5,4,4,4,5,4) var.test(A,B) # Test sur la variance t.test(A, B, paired = FALSE, var.qual = TRUE)
Test unilatéral droit:
t.test(A, B, paired = FALSE, var.qual = TRUE, alternative = "greater")
Test unilatéral gauche:
t.test(A, B, paired = FALSE, var.qual = TRUE, alternative = "less")
Ce test nécessite la normalité des données dans chaque sous-population. Vous pouvez la tester avec le code suivant :
shapiro.test(A) shapiro.test(B)
Test de T sur deux échantillons indépendants avec variance différente
La fonction t.test() permet – entre d’autres – de comparer deux moyennes issues de deux échantillons indépendants avec variances différentes (Test de Welch).
Test bilatéral:
C <- c(2,4,5,4,2,3,5,4,6,4,2,7,1,2,1,3) D <- c(5,6,4,5,3,5,6,5,5,4,4,5,4) var.test(C,D) # Test sur la variance t.test(C, D, paired = FALSE, var.qual = FALSE)
Test unilatéral droit:
t.test(C, D, paired = FALSE, var.qual = FALSE, alternative = "greater")
Test unilatéral gauche:
t.test(C, D, paired = FALSE, var.qual = FALSE, alternative = "less")
Ce test nécessite la normalité des données dans chaque sous-population. Vous pouvez la tester avec le code suivant :
shapiro.test(C) shapiro.test(D)
Test de T sur deux échantillons dépendants
La fonction t.test() permet – entre d’autres – de comparer deux moyennes issues de deux échantillons dépendants.
Test bilatéral:
E <- c(3,4,5,4,3,4,4,4,5,3,3,6,1,2) F <- c(5,7,6,4,5,3,5,5,3,6,5,5,4,4) t.test(E, F, paired = TRUE)
Test unilatéral droit:
t.test(E, F, paired = TRUE, alternative = "greater")
Test unilatéral gauche:
t.test(E, F, paired = TRUE, alternative = "less")
Ce test nécessite la normalité dans les scores des différences:
shapiro.test(F-E)
ANOVA à un critère de classification
La fonction lm() permet de créer des modèles linéaires. De ce fait, elle peut être utilisée pour construire des modèles ANOVA.
data(InsectSprays) boxplot(InsectSprays$count ~ InsectSprays$spray) LinMod1 <-lm(InsectSprays$count ~ InsectSprays$spray) summary(LinMod1) # Paramètres du modèle anova(LinMod1) # Tableau de l’ANOVA
Condition d’application : normalité des résidus et homoscédasticité
par(mfrow = c(2,2)) plot(LinMod1, col.smooth = "black") # Analyse des résidus bartlett.test(InsectSprays$count ~ InsectSprays$spray) # Test sur l’homogénéité des variance de Bartlett library(car) leveneTest(InsectSprays$count ~ InsectSprays$spray) # Test sur l’homogénéité des variance de Levene. Pour utiliser cette fonction il faut installer le package « car ».
ANOVA à deux critères de classification
La fonction lm() permet de créer des modèles linéaires. De ce fait, elle peut être utilisée pour construire des modèles ANOVA :
data(CO2) boxplot(CO2$uptake ~ CO2$Treatment * CO2$Type) interaction.plot(CO2$Treatment, CO2$Type, CO2$uptake, main = "Graphique des moyennes", xlab = "Traitement", ylab = "Consommation de CO2", trace.label = "Provenance")
Modèle avec interaction (multiplicatif) :
LinMod2 <- lm(CO2$uptake ~ CO2$Treatment * CO2$Type) # Construction du modèle avec interaction (multiplicatif) summary(LinMod2) # Paramètres du modèle anova(LinMod2) # Tableau de l’ANOVA
Condition d’application : normalité des résidus et homoscédasticité
par(mfrow = c(2,2)) plot(LinMod2, col.smooth = "black") # Analyse des résidus bartlett.test(CO2$uptake ~ CO2$Treatment * CO2$Type) # Test sur l’homogénéité des variance de Bartlett library(car) leveneTest(CO2$uptake ~ CO2$Treatment * CO2$Type) # Test sur l’homogénéité des variance de Levene. Pour utiliser cette fonction il faut installer le package « car ».
Graphes et réseaux
Il y a deux packages sur R permettant de générer et analyser des réseaux : igraph et statnet. Ces deux outils présentent des avantages et des désavantages :
- igraph permet de représenter les réseaux de manière efficace et élégante. De plus, il s’agit d’un outil intéressant pour génération aléatoire des graphes (simulation).
- Contrairement à igraph, statnet permet de faire de la modélisation statistique (voir Exponential random graph models – ERGP).
Nous nous limiterons ici à présenter statnet.
Générer un graphe
Pour créer un graphe, il faut avant tout créer une matrice d’adjacence ou une matrice des arrêts. Générons une matrice d’adjacence avec le code suivant :
mat_adjacence <- matrix(round(runif(2500, max = 0.51)), ncol = 50)
La matrice des arêtes peut être crée de la manière suivante :
depart <- c('A', 'A', 'A', 'A', 'A', 'B', 'B', 'B', 'C', 'C', 'D', 'D', 'D', 'D', 'D', 'F', 'G', 'G')
arrive <- c('B', 'C', 'D', 'F', 'G', 'A', 'C', 'G', 'F', 'G', 'A', 'B', 'C', 'F', 'G', 'H', 'B', 'C')
mat_aretes <- cbind(depart,arrive)
La fonction as.network() permet de transformer une matrice d’adjacence ou une matrice des arrêts en un objet de type network (un graphe). Pour utiliser cette fonction, il faut charger la librarie statnet :
library(statnet) G1 <- as.network(mat_adjacence) G2 <- as.network(mat_aretes)
Alternativement, nous pouvons générer un graphe aléatoire en définissant le nombre de sommets et sa densité :
G3 <- rgraph(20, tprob = 0.1) # G3 est un graphe aléatoire de 20 sommets, avec densité 0.1
Représenter un graphe
Les fonctions gplot et gplot3d permettent de générer des graphiques en 2 et 3 dimensions :
gplot(G1) gplot3d(G1) gplot(G2) gplot3d(G2) gplot(G3) gplot3d(G3)
La fonction plot.sociomatrix() permet de représenter de manière efficace la matrice d'adjacence d'un graphe :
plot.sociomatrix(G2)
Indices, indicateurs
La librairie statnet permet de calculer des indicateurs permettant de décrire les réseaux et leurs éléments.
Au niveau du réseau
La fonction gden permet d’obtenir la densité d’un reseau :
gden(G1) gden(G2) gden(G3)
La fonction grecip() permet de calculer le degré de réciprocité d'un réseau :
grecip(G1) grecip(G2) grecip(G3)
La fonction gtrans() permet d’estimer le niveau de transitivité d’un graphe donné :
gtrans(G1) gtrans(G2) gtrans(G3)
La fonction connectedness d’éstimer le degré de connexité d’un réseau :
connectedness(G1) connectedness(G2) connectedness(G3)
Au niveau des sommets
La fonction degree() permet d’obtenir le degré de chaque sommet du graphe. L’argument cmode permet de spécifier si on s’intéresse aux degrés entrants (indegree), sortants (outdegree), ou totaux (freeman) :
degree(G1, cmode = "indegree") degree(G1, cmode = "outdegree") degree(G1, cmode = "freeman")
degree(G2, cmode = "indegree") degree(G2, cmode = "outdegree") degree(G2, cmode = "freeman")
degree(G3, cmode = "indegree") degree(G3, cmode = "outdegree") degree(G3, cmode = "freeman")
Lorsqu’on étude un réseau comportant un grand nombre de sommets, on peut étudier le tri à plat des degrés :
table(degree(G1, cmode = "indegree")) table(degree(G1, cmode = "outdegree")) table(degree(G1, cmode = "freeman"))
La centralité de proximité peut être mesurée à partir de la fonction closeness() :
closeness(G1) closeness(G2) closeness(G3)
La fonction betweenness() permet d’obtenir la centralité d’intermédiarité :
betweenness(G1) betweenness(G2) betweenness(G3)
infocent() permet d’évaluer le niveau de centralité de chaque sommet vis-à-vis de la circulation de l’information :
infocent(G1) infocent(G2) infocent(G3)
Les indices de closeness(), de betweenness(), infocent() peuvent être représentées sur la représentation du réseau :
gplot(G2, vertex.cex = closeness (G2) * 3, main = " closeness ") gplot(G2, vertex.cex = betweenness(G2) * 0.6, main = "betweenness") gplot(G2, vertex.cex = infocent (G2) * 1.3, main = " infocent ")
La fonction geodist()$count permet de compter le nombre de chemins possibles entre chaque paire de sommets du réseau :
geodist(G2)$count
La fonction geodist()$gdist indique la distance entre chaque paire de sommets du sommet. La distance se définie par la longueur d’un plus court chemin.
geodist(G2)$gdist
Packages, bibliographie et liens
Learning Analytics Moodle
cereghd0 a créé des fonctions permettant d'analyser les rapports de Moodle concernant l'activité des utilisateurs : Learning Analytics Moodle. Ce logiciel est destiné aux enseignants et aux chercheurs en technologies éducatives.
Packages utiles
- Rcmdr : interface graphique de R , http://socserv.mcmaster.ca/jfox/Misc/Rcmdr/
- OpenMx : outil permettant de générer et évaluer des modèles à équations structurales , http://openmx.psyc.virginia.edu/
- igraph : outil permettant de créer et analyser des graphes et des réseaux , http://igraph.sourceforge.net/
- statnet : outil permettant de créer et analyser des graphes et des réseaux , http://statnet.csde.washington.edu/
- lattice : outil permettant de générer des représentations graphiques avancées , http://lmdvr.r-forge.r-project.org/figures/figures.html
- R.matlab : outil permettant d’importer des fichiers Matlab sur R , http://cran.r-project.org/web/packages/R.matlab/R.matlab.pdf
- R2HTML : outil permettant d'exporter les outputs de R en HTML , http://cran.r-project.org/web/packages/R2HTML/R2HTML.pdf
- corrgram : outil permettant de représenter graphiquement des matrices de corrélations , http://cran.r-project.org/web/packages/corrgram/corrgram.pdf
- 3dscatterplot : outil permettant à créer des graphiques 3D , http://cran.r-project.org/web/packages/scatterplot3d/scatterplot3d.pdf
- lavaan: latent variable analysis
- semPlot: outil pour visualiser des modèles SEM, http://cran.r-project.org/web/packages/semPlot/index.html
Pour les outils text mining, voir text mining avec R
Liens
Manuels et résumés
- R Reference Card. Résumé officiel des commandes R.
- Yanchang Zhao's R Reference Card for Data Mining, aussi disponible à http://www.rdatamining.com/
Sites R
(tutoriels, exemples, etc.)
- Tutoriels R de E.Horber (UniGE)
- Statmethods.net (Quick-R)
- RDM
- Introduction to R (Tutoriel avec exerices). Demande de s'enregistrer
Bibliographie
- Adler, J. (2011). R, L’essentiel. Pearson.
- Beauguitte, L. (2012). Analyser les réseaux avec R (packages statnet, igraph et tnet). http://cel.archives-ouvertes.fr/docs/00/68/78/71/PDF/fmr12_analyse_de_graphe_avec_R.pdf
- Barnier, J. (2011). L'analyse des réseaux avec R. http://alea.fr.eu.org/git/doc_reseaux_r.git/blob_plain/HEAD:/networks.pdf
- Chevalier, B. (2005). Logiciels libres Open source : qu'est-ce que c'est ? Paris : H & K.
- Cornillon, P.A. (2010). Statistiques avec R (2ème édition augmentée). Rennes : Presses Universitaires de Rennes.
- Howell, D. (1998). Méthodes statistiques en sciences humaines. Bruxelles : Editions De Boeck Université.
- Huillet, J. Initiation à l’environnement R. http://cict.fr/~stpierre/doc-R.pdf
- Jean, B. (2011). Du bon usage des licences libres. Framasoft (coll. Framabook).
- Millot, G. (2008). Comprendre et réaliser les tests statistiques à l’aide de R (1ère édition). Bruxelles : Editions De Boeck Université.
- Smets-Solanes, J.P. (1999). Logiciels libres : liberté, égalité, business. Paris : Edispher.
Cereghd0 9 janvier 2013 à 12:27 (CET)