« Gènes constitués d'Introns et d'Exons » : différence entre les versions
Aller à la navigation
Aller à la recherche
(bases du protocole) |
m (introduction d'une image mise au point du texte) |
||
| Ligne 3 : | Ligne 3 : | ||
== Procédure == | == Procédure == | ||
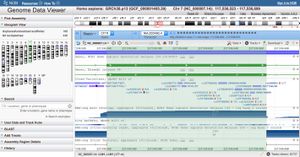
Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici] | Ouvrir Genome data Viewer GDV [https://www.ncbi.nlm.nih.gov/genome/gdv/ ici] | ||
[[Fichier:CFTR gene Exon7 highlith in GDV.jpg|alt=CFTR gene Exon7 highlith in GDV|vignette|CFTR gene Exon7 highlith in GDV]] | |||
Sélectionner un gène (p. ex CFTR) selon protocole | Sélectionner un gène (p. ex CFTR) selon protocole | ||
Cliquer les petits ronds dans la bande bleue pour mettre en évidence un exon. | |||
Observer la séquence d'ADN correspondante et le sens de lecture ( flèches dans la bande verte) | |||
Observer les bandes bleues plus bas sous RNAseq exon coverage et RNAseq intron spanning read | |||
Puis un autre | |||
== Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe == | == Ce qu'on peut obtenir : p. ex, synthèse par un élève des résultats avec une classe == | ||
Version du 13 mars 2020 à 18:17
Éprouver que certains gènes sont constitués d'Introns et d'Exons
Procédure
Ouvrir Genome data Viewer GDV ici
Sélectionner un gène (p. ex CFTR) selon protocole
Cliquer les petits ronds dans la bande bleue pour mettre en évidence un exon.
Observer la séquence d'ADN correspondante et le sens de lecture ( flèches dans la bande verte)
Observer les bandes bleues plus bas sous RNAseq exon coverage et RNAseq intron spanning read
Puis un autre