« Preuve de l'évolution par la comparaison de protéines chez différentes espèces » : différence entre les versions
m (rédaction) |
m (clean un edit) |
||
| Ligne 28 : | Ligne 28 : | ||
=== Produire un alignement === | === Produire un alignement === | ||
Cliquer le bouton Align en-dessus du bandeau en haut de la liste. Après un temps | Cliquer le bouton "Align" en-dessus du bandeau en haut de la liste. Après un temps (plusieurs secondes, voire minutes) l'alignement apparait. | ||
[[Fichier:Alignement-ins-similarity.jpg|alt=Alignement de quelques insulines de diverses espèces - similarité activée |vignette|Alignement de quelques | [[Fichier:Alignement-ins-similarity.jpg|alt=Alignement de quelques insulines de diverses espèces - similarité activée |vignette|Alignement de quelques CFTR de diverses espèces - similarité activée ]] | ||
Les séquences alignées sont affichées sous forme de tableau avec 60 a.a. par ligne, une étoile "*" signifie '''identité''' ( dans la colonne) pour '''toutes les séquences alignées'''. ":" signifie acides aminées qui ont des propriétés physico-chimiques '''très''' similaires, "." signifie acides aminées qui ont des propriétés physico-chimiques '''similaires''', un "-" signifie que le le programme d'alignement a introduit un espace ("gap") pour aligner avec des séquences plus longues. | |||
Pour retrouver le nom de l'espèce, cliquer sur le numéro d'accession en bleu -> ouvre la fenêtre de cette protéine avec le nom complet de l'espèce. | |||
'''Pour aller plus loin''' | '''Pour aller plus loin''' | ||
Version du 27 mars 2020 à 18:27
Preuve de l'évolution par la comparaison de séquences de protéines chez différentes espèces
Procédure
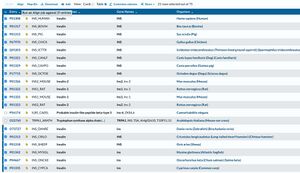
Trouver les entrées pour insuline de plusieurs espèces dans la banque de données UniProtKB
UniProtKB contient toutes les protéines répertoriées à partir des données publiques (~180 millions début 2020).
N. B. : En principe on utilise les séquences protéiques : elles sont plus pertinentes du point de vue évolutif (phénotype). Ici nous travaillons avec la CFTR qui cause la mucoviscidose quand elle est défectueuse, mais d'autres protéines peuvent être employées (cf liste d'exemples)
Aller sur UniProtKB dans Query taper CFTR. Il faut taper le nom du gène ( même si c'est une base de protéines : )
Uniprot va chercher toutes les entrées dans lesquelles on trouve le terme CFTR solution : la liste est énorme.
Cliquer Restrict term to ....Protein name et cliquer Filter by puis "reviewed" solution (Insuline) , solution CFTR NB: le gène a un synonyme abcc7
N.B. Le nom de la protéine (cf liste d'exemples) diffère souvent du nom du gène. Il vaut mieux chercher par le nom du gène pour retrouver une protéine. UniProtKB est une banque de données sur les protéines : les informations sont focalisées sur les séquences protéiques mais la liste des noms de gènes est toutefois exhaustive.
Sélectionner la même protéine chez plusieurs organismes
Sélectionner (cocher) dans cette liste les insulines des espèces qui vous intéressent. Un minimum de 5 est nécessaire si l'on veut ensuite former un alignement de séquences raisonnable. (cf liste d'exemples)
Afficher un grand nombre d'entrées en modifiant "Show" en bas du tableau ( p. ex 50, voire 100)
Les séquences son surlignées de jaune et leur nombre dans le bandeau jaune en haut. Avant de passer à la page suivante cliquer Add to basket sinon on perd la sélection.
(N.B: si on avait sélectionné d'autres séquences auparavant : il faut cliquer Clear dans ce bandeau pour éviter qu'elles restent sélectionnées.)
Si on veut passer à la page suivante il faut mettre ces séquences dans le panier
Produire un alignement
Cliquer le bouton "Align" en-dessus du bandeau en haut de la liste. Après un temps (plusieurs secondes, voire minutes) l'alignement apparait.
Les séquences alignées sont affichées sous forme de tableau avec 60 a.a. par ligne, une étoile "*" signifie identité ( dans la colonne) pour toutes les séquences alignées. ":" signifie acides aminées qui ont des propriétés physico-chimiques très similaires, "." signifie acides aminées qui ont des propriétés physico-chimiques similaires, un "-" signifie que le le programme d'alignement a introduit un espace ("gap") pour aligner avec des séquences plus longues.
Pour retrouver le nom de l'espèce, cliquer sur le numéro d'accession en bleu -> ouvre la fenêtre de cette protéine avec le nom complet de l'espèce.
Pour aller plus loin
Cocher la case "Similarity" dans la colonne de gauche : on peut observer que certaines zones sont plus conservées.
Cocher la case "Transmembrane" : on peut observer des zones susceptibles d'être transmembranaires
Cocher la case "Natural Variant" : on peut observer des zones susceptibles de différer dans la population humaine (SNP, … )
Cocher la case "DNA Binding" : Lorsque c'est pertinent, on peut observer des zones susceptibles de se lier à l'ADN
Compléments
Les propriétés chimiques des différents a.a. sont décrites ici
Un tableau de correspondance des codes à 3 lettres et à1 lettre et codons accessible ici .
Un arbre phyogénétique ?
L'arbre produit en dessous est un 'guided tree' utilisé par le programme pour construire l'alignement, construit seulement sur la base des différences entre les séquences. Ce n'est pas un arbre phylogénétique plus complexe à établir.