Approche moléculaire
Phylogénie moléculaire
Deux domaines d'application majeurs :
- Reconstruire l'histoire évolutionnaire de taxons, caractères ou de gènes.
- Analyse de caractères et de vitesses d'évolution
La phylogénie permet d'étudier et comprendre comment ces objets biologiques ont changé au cours du temps. C'est la première étape d'une étude
comparative.
Définition :
C'est la reconstruction de l'histoire évolutionnaire d'objets biologiques (organismes, gènes …) par comparaison de séquences d'acides nucléiques ou protéiques. L'hypothèse de travail est plus des objets partagent un ancêtre proche, plus leurs séquences doivent se ressembler.
Pour effectuer cette analyse, il faudra donc que les séquences comparées soient orthologues (même gène chez différentes espèces) et il vaudra mieux que la pression de sélection soit plus ou moins restée la même au cours du temps dans les différentes lignées.
Exemple :
- La comparaison des séquences de protéines.
- Cytochrome C ou B, actine, collagène, Histone, ...
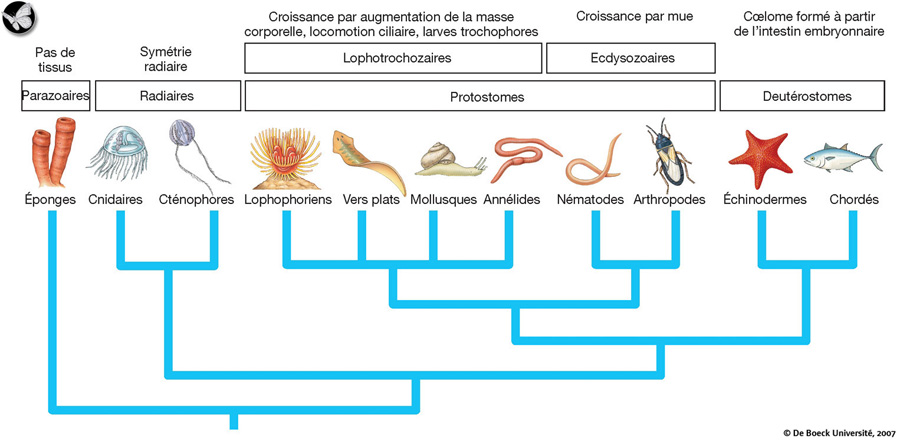
Les Animaux sont classés traditionnellement en quelques 35 phylums.
Jusqu'il y a peu, on a établi les relations évolutives entre les phylums animaux en supposant que les phylums sont apparentés lorsqu'ils partagent certaines caractéristiques morphologiques fondamentales, que l'on présume être apparue une seule fois.
Phylogénie traditionnelle des protostomes: les biologistes ont séparé traditionnellement
les animaux à symétrie bilatérale en trois groupes qui diffèrent par leur cavité corporelle: les
acoelomates, les pseudocoelomates et les coelomates.
Cette phylogenèse traditionnelle est à présent réexaminée. En effet, la construction de phylogénies à partir de données moléculaires est en passe de modifier de manière significative notre perception des relations entre les phylums animaux. En effet, la systématique moléculaire utilise des séquences uniques dans certains gènes afin d'identifier des ensembles de groupes apparentés.
Phylogénie des protostomes basée sur l'ARNr: les biologistes ont séparé traditionnellement les animaux à symétrie bilatérale en trois groupes qui diffèrent par leur cavité corporelle: les acoelomates, les pseudocoelomates et les coelomates.
Un défi majeur de la biologie évolutive moderne est donc de trouver le moyen de lier l'évolution des séquences d'ADN avec l'évolution des caractères morphologiques complexes que l'on a utilisé pour élaborer la phylogénie traditionnelle.
Comparer les génomes de différentes espèces fournit un nouvel outil puissant pour explorer ces relations. Cependant, les phylogénies reposant sur des molécules différentes suggèrent parfois des relations évolutives tout à fait différentes, ce qui complique bien évidemment l'analyse.
A lire l'article : "Retracer l'histoire évolutive" LA RECHERCHE n°415 - Janvier 2008
Sites utilisés :
Uniprot : Obtention des séquences de protéines
Clustal W : Comparaison des séquences
Phylodendron : Construction des arbres phylogénétiques.
Wikipédia Encyclopédie libre
Marche à suivre
- Avec l’aide de l’encyclopédie Wikipédia, sélectionner un organisme par embranchement (Exemple : Pieuvre).
- Obtenir le Genre (Octopus).
- Copier cette information.
- Ouvrir le site Uniprot. Choisir dans le menu search in Taxonomy.
- Dans Query coller le genre de l’organisme choisi.
- Choisir les résultats possédant une étoile et les organismes les plus communs (Octopus vulgaris).
- Sélectionner UniProtKB.
- Dans Query ajouter à taxonomy:6645 AND Cytochrome c oxidase subunit 1.
- Cliquer sur le numéro d’Accession.
- Cliquer sur le rectangle orange FASTA.
- Copier les séquences obtenues et coller dans le Bloc-Notes.
- Répéter les points 1 à 4 pour chaque organisme et coller à la suite les séquences obtenues dans le Bloc-Notes.
- Organismes:
- 1 spongiaire (porifère)
- 1 cnidaire (méduse, corail, ...)
- 1 échinoderme (oursin, étoile de mer)
- 2 vers (plat, rond, annelé)
- 2 mollusques (gastéropode, céphalopode, bivalve)
- 2 arthropodes (insecte. arachnide, crustacé, myriapode)
- 3 vertébrés (poisson, amphibien, reptile, oiseau) et 2 mammifères.
- Sauvegarder le fichier.
- Sauvegarder le fichier.
- Ouvrir le site Clustal W.
- Copier l'ensemble des données du Bloc-Notes (ctrl-A).
- Coller le tout dans le rectangle en bas de la page.
- Presser run.
- Dans le rectangle Results of search, sélectionnez Guide tree file.
- Copier la totalité de l'information.
- Ouvrir le site Phylodendron.
- Coller l'information dans le rectangle.
- Choisir le style d'arbre (Tree styles).
- A ce stade vous pouvez modifier le texte et écrire le nom complet des animaux à la place des codes proposés.
- Exemple : (P00395|COX1_HUMAN:0.005848,Q9T9X0|COX1_PANPA:0.005848); devient (P00395|Homme:0.005848,Q9T9X0|Bonobo:0.005848);
- Choisir le Output Format soit PDF ou GIF Image Map.
- Presser Submit
- Enregistrer le fichier
- Exemple d'arbre obtenu en comparant le cytochrome b :