Approche moléculaire
Aller à la navigation
Aller à la recherche
Sites utilisés :
Uniprot : Obtention des séquences de protéines
Clustal W : Comparaison des séquences
Phylodendron : Construction des arbres phylogénétiques.
Wikipédia Encyclopédie libre
Marche à suivre
- Avec l’aide de l’encyclopédie Wikipédia, sélectionner un organisme par embranchement (Exemple : Pieuvre).
- Obtenir le Genre (Octopus).
- Copier cette information.
- Ouvrir le site Uniprot. Choisir dans le menu search in Taxonomy.
- Dans Query coller le genre de l’organisme choisi.
- Choisir les résultats possédant une étoile et les organismes les plus communs (Octopus vulgaris).
- Sélectionner UniProtKB.
- Dans Query ajouter à taxonomy:6645 AND Cytochrome c oxidase subunit 1.
- Cliquer sur le numéro d’Accession.
- Cliquer sur le rectangle orange FASTA.
- Copier les séquences obtenues et coller dans le Bloc-Notes.
- Répéter les points 1 à 4 pour chaque organisme et coller à la suite les séquences obtenues dans le Bloc-Notes.
- Organismes:
- 1 spongiaire (porifère)
- 1 cnidaire (méduse, corail, ...)
- 1 échinoderme (oursin, étoile de mer)
- 2 vers (plat, rond, annelé)
- 2 mollusques (gastéropode, céphalopode, bivalve)
- 2 arthropodes (insecte. arachnide, crustacé, myriapode)
- 3 vertébrés (poisson, amphibien, reptile, oiseau) et 2 mammifères.
- Sauvegarder le fichier.
- Sauvegarder le fichier.
- Ouvrir le site Clustal W.
- Copier l'ensemble des données du Bloc-Notes (ctrl-A).
- Coller le tout dans le rectangle en bas de la page.
- Presser run.
- Dans le rectangle Results of search, sélectionnez Guide tree file.
- Copier la totalité de l'information.
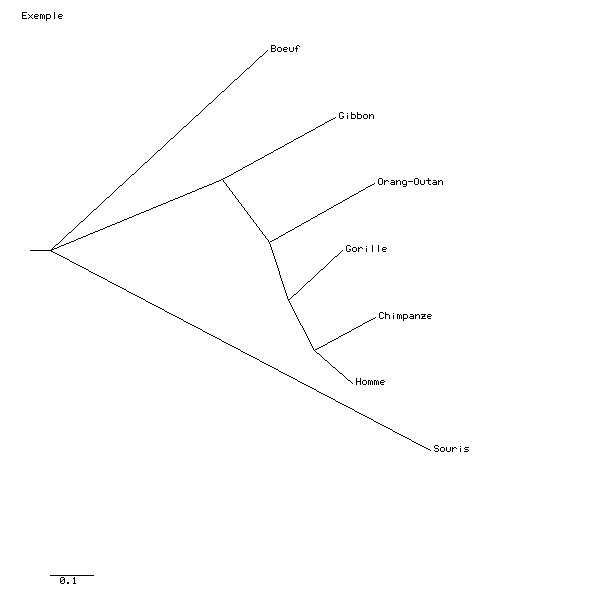
- Ouvrir le site Phylodendron.
- Coller l'information dans le rectangle.
- Choisir le style d'arbre (Tree styles).
- A ce stade vous pouvez modifier le texte et écrire le nom complet des animaux à la place des codes proposés.
- Exemple : (P00395|COX1_HUMAN:0.005848,Q9T9X0|COX1_PANPA:0.005848); devient (P00395|COX1_Homme:0.005848,Q9T9X0|COX1_Bonobo:0.005848);
- Choisir le Output Format soit PDF ou GIF Image Map.
- Presser Submit
- Enregistrer le fichier
- Résultat :